
Obliczenia w biochemii i biologii molekularnej
Obliczenia w biochemii i biologii molekularnej
Do przeprowadzenia reakcja PCR potrzeba:
1. Jednoniciowej matrycy DNA.
2. Pary starterów (sekwencji oligonukleotydów komplementarnych do końców
zdefiniowanej sekwencji matrycowego DNA).
3. Trójfosforanów deoksyrybonukleotydów (dNTP).
4. Enzymu polimerazy DNA.
Każdy cykl reakcji składa się z trzech etapów:
1.
Termiczna denaturacja
powielanego DNA (temp. 94 - 95°C) w obecności
nadmiaru każdego z dwóch starterów i czterech dNTP.
2.
Hybrydyzacja starterów
do sekwencji komplementarnej z matrycą w
temperaturze 40 - 60°C przez 20 do 40 sekund.
3.
Wydłużanie startera
od końca 3'-OH przez sukcesywne dosyntetyzowanie
dNTP w temperaturze 72°C. Proces ten jest katalizowany przez polimerazę DNA.
PCR – podsumowanie

Obliczenia w biochemii i biologii molekularnej
Powtarzanie cykli amplifikacji prowadzi do wzrostu ilości
odpowiednich fragmentów DNA w postępie wykładniczym,
2
n
gdzie n oznacza liczbę cykli
Po 30 - 40 cyklach uzyskuje się powielenie fragmentu DNA
kilka milionów razy - produkty jednego cyklu amplifikacji
służą jako matryce dla następnego.
Główny produkt tej eksponencjalnej reakcji to segment
dwuniciowego DNA , którego końce określone są prze końce
5' primera i których długość określona jest przez odległość
pomiędzy primerami.
PCR – podsumowanie

Obliczenia w biochemii i biologii molekularnej
Na efektywność reakcji PCR w istotny sposób
wpływają różne czynniki fizyczne i chemiczne.
Czynnikami takimi są:
1. Typ termocyklera.
2. Temperatura i czas poszczególnych etapów,
ilość cykli.
3. Stężenie polimerazy DNA, jakość enzymu i
buforu reakcyjnego.
4. Stężenie dNTP.
5. Primery.
Optymalizacja warunków reakcji PCR

Obliczenia w biochemii i biologii molekularnej
Optymalizacja warunków reakcji PCR
Temperatura i czas poszczególnych etapów
Denaturacja – typowe warunki dla niemal każdej reakcji z
polimerazą Taq to 95°C 20 - 30 sekund, lub 97°C - 15 sekund.
Wyższa temperatura może być stosowana w przypadku matrycy
bogatej w pary G+C.
Niekompletna denaturacja może obniżyć wydajność reakcji – za mało
matrycy w kolejnych cyklach.
Za długa denaturacja może doprowadzić do niepotrzebnego
zmniejszenia aktywności enzymu.
Czas aktywności polimerazy Taq: >2 godz. przy 92,5°C, 40 minut
przy 95°C i 5 minut dla temperatury 97,5°C.
Denaturacja nie powinna być ostatnim etapem kończącym proces
amplifikacji PCR.

Obliczenia w biochemii i biologii molekularnej
Optymalizacja warunków reakcji PCR
Temperatura i czas poszczególnych etapów
Hybrydyzacja (ang. annealing)– przyłączenie startera, najbardziej istotny czynnik w optymalizacji
reakcji.
Na ten etap ma wpływ a) temperatura, b) stężenie matrycy i startera oraz c) czas.
Po denaturacji mieszaninę reakcyjną schładza się do około 50 – 65°C w której startery mogą łatwo i
dokładnie hybrydyzować z matrycą. Czas hybrydyzacji – 20 – 40 sekund.
Jeżeli temperatura hybrydyzacji jest za wysoka – przyłączenie starterów jest nie możliwe.
Przy za niskiej temperaturze może zachodzić niespecyficzne przyłączenie starterów do wielu miejsc
na matrycy – w efekcie duża ilość nie specyficznych produktów o nie znanej sekwencji.
Temperaturę hybrydyzacji należy ustalać oddzielnie dla każdego startera, można to już przewidzieć na
etapie projektowania. Ważnym parametrem do określenia jest temperatura topnienia Tm (malting
temperature) zaprojektowanej struktury.
Jeśli temperatura topnienia będzie wyższa niż temperatura hybrydyzacji to z reakcji nic nie będzie.
Nie dojdzie do hybrydyzacji a to jest warunek rozpoczęcia reakcji przez polimerazę.
Jeśli Tm jest niższa niż temperatura hybrydyzacji nic nie powinno zakłócić reakcji. Wybiera się
najwyższą możliwą temperaturę hybrydyzacji, znacząco wpływa to na specyficzność reakcji.
Na temperaturę hybrydyzacji wpływa budowa primera, a dokładnie procentowa zawartość par G+C
w stosunku do par AT oraz jego długość.

Optymalizacja warunków reakcji PCR
Temperatura i czas poszczególnych etapów
Obliczenia w biochemii i biologii molekularnej
Elongacja -kiedy startery połączą się specyficznie z matrycą do
pracy przystępuje polimeraza.
Standardowo dla polimerazy Taq reakcję polimeryzacji
przeprowadza się przy 72°C przez 20 sekund - dla fragmentów
krótszych niż 500 pz i 40 sekund - dla fragmentów o długości
do 1,2 kpz.
W przypadku innych polimeraz należy się kierować
wskazaniami producenta odnośnie temperatury i czasu
elongacji
Zakończenie amplifikacji – 4 °C

Optymalizacja warunków reakcji PCR
Temperatura i czas poszczególnych etapów
Obliczenia w biochemii i biologii molekularnej
Ilość cykli:
●
Zależy od początkowego stężenia matrycy DNA.
●
Nie powinna przekraczać 40 (optymalna od 25 do 35).
●
Zwiększając liczbę cykli wzrost niespecyficznych produktów.
●
Zbyt mała liczba cykli mała wydajność powstającego produktu.

Obliczenia w biochemii i biologii molekularnej
Optymalizacja warunków reakcji PCR
Stężenie polimerazy DNA
Optymalna ilość enzymu wynosi 0,5 - 2,5 jednostki (U) w próbce na
50 μl na około 40 cykli.
●
Zwiększenie stężenia enzymu - niespecyficzne produkty
●
Zmniejszenie stężenia enzymu - zbyt małe ilości produktu.

Obliczenia w biochemii i biologii molekularnej
Optymalizacja warunków reakcji PCR
Skład buforu reakcyjnego
Stężenie jonów Mg
2+
●
Optymalne stężenie około l,5 mM, (0,5 – 5 mM)
●
Jony Mg
2+
wpływają na aktywność enzymu, zwiększają Tm ds DNA,
tworzą rozpuszczalne kompleksy z dNTP oraz z powstającymi z nich w
trakcie polimeryzacji pirofosforanem oraz EDTA
●
Zbyt wysokie stężenie Mg
2+
niespecyficzne produkty
●
Zbyt niskie stężenie Mg
2+
- słabszy sygnał amplifikacji.

Obliczenia w biochemii i biologii molekularnej
Optymalizacja warunków reakcji PCR
Stężenie dNTP
Optymalne stężenie dNTP długość reakcji PCR zależy od:
●
Długości amplifikowanego produktu,
●
Stężenia MgCl
2
●
Stężenia primera
Końcowe stężenie każdego nukleotydu w roztworze wynosi około 200 μM
ale zależy od rodzaju polimerazy i prowadzonej reakcji.
Nierówne ilości czterech dNTP redukują wydajność amplifikacji.
Nukleotydy (i pirofosforan) redukują pulę wolnych jonów Mg
2+
(wpływając na aktywność polimerazy i hybrydyzację starterów).
Niskie stężenie dNTP minimalizuje błędne ich wstawianie w nowo
syntetyzowanej nici.

Obliczenia w biochemii i biologii molekularnej
Optymalizacja warunków reakcji PCR
Startery
●
Długość typowych starterów powinna wynosić 18 do 28 nukleotydów,
zawierających 50 - 60 % par G-C.
●
Końcowe optymalne stężenie starterów wynosi 0,1 do 0,5 μM wyższe
stężenie może prowadzić do akumulacji niespecyficznych produktów i
zwiększa prawdopodobieństwo generowania dimerów starterów.
●
3' końce starterów powinny być niekomplementarne w stosunku do siebie
(niebezpieczeństwo tworzenia dimerów).
●
Nie powinny zawierać sekwencji palindromicznych.
●
Nie powinny tworzyć struktur drugorzędowych.
●
Należy unikać w sekwencjach starterów nierównej dystrybucji rejonów
bogatych w G/C lub A/T.
●
Ważne jest, aby Tm obydwu starterów były do siebie zbliżone, najlepiej
takie same.
●
Tm nie powinna być niższa niż 50°C.

Obliczenia w biochemii i biologii molekularnej
Projektowanie starterów
Temperatura stapiania starterów
●
Najprostszy wzór na obliczanie temperatury stapiania (stosowany
najczęściej):
T
m
= 2°(A + T) + 4°(G +C)
Jest prawdziwy dla oligonukleotydów o długości 14-20 pz. Dla dłuższych
starterów wprowadza się korekcję temperatury obniżając ją o 5-10°C
●
Obliczenia oparte o stężenie soli, zawartość par G-C i długość startera:
L – długość startera
[SALT]- zawartość soli w buforze reakcyjnym:
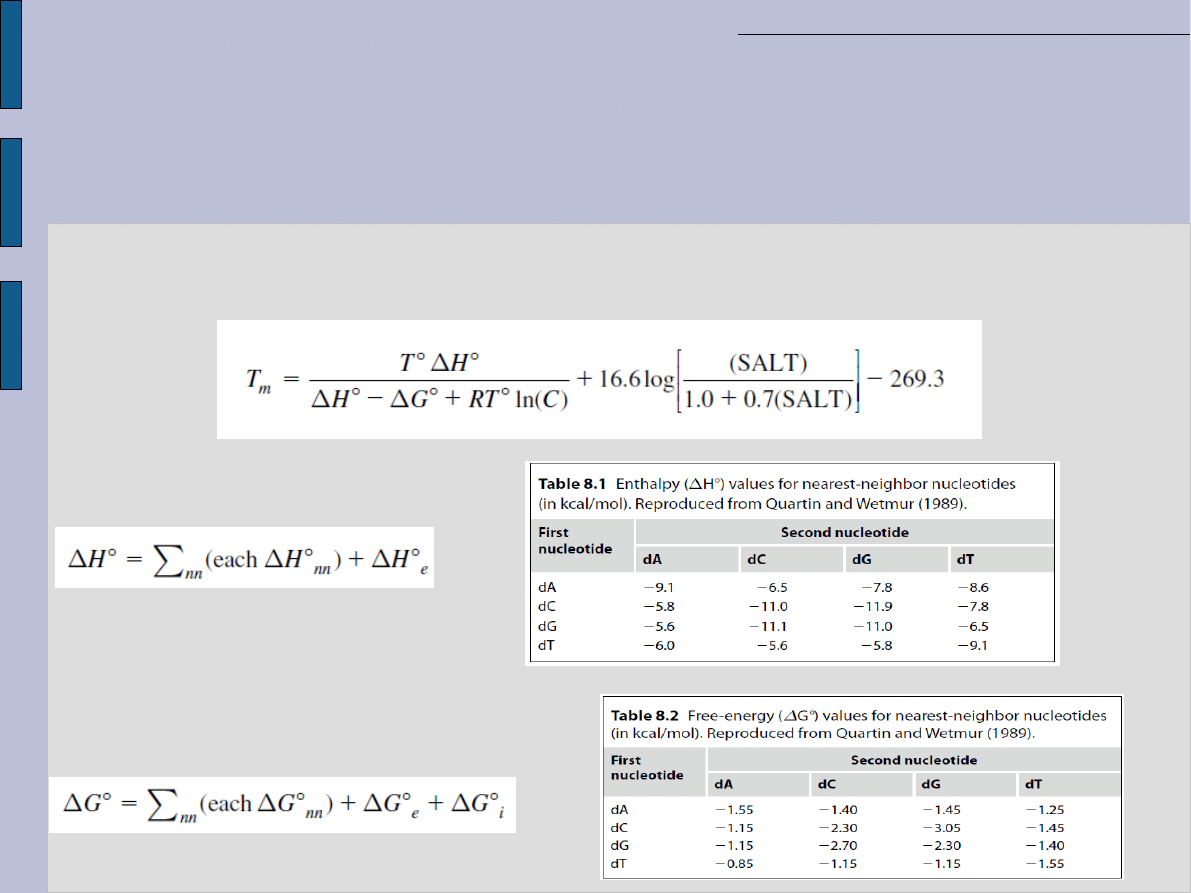
Projektowanie starterów
Temperatura stapiania starterów
Projektowanie starterów
Temperatura stapiania starterów
Obliczenia w biochemii i biologii molekularnej
●
Obliczenia oparte o oddziaływania między nukleotydami w najbliższym
sąsiedztwie:
Wartości entalpii
ΔH
o
e
=-5kcal/mol
Wartości energii swobodnej
ΔG
o
e
=-1kcal/mol, ΔG
o
i
=+2,2kcal/mol

Projektowanie starterów
Długość starterów
Obliczenia w biochemii i biologii molekularnej
W standardowym PCR stosuje się startery o długości 18-24 pz.
Minimalna długość (18pz) wynika z wielkości genomu i
prawdopodobieństwa spotkania w sekwencji 2 identycznych
fragmentów (specyficzność oddziaływania starter-matryca).
Z każdym kolejnym nukleotydem starter staje się 4 x bardziej
specyficzny.
Im dłuższy starter tym mniejsza wydajność reakcji amplifikacji, bo
trudniej podczas stapiania utworzyć prawidłowy dupleks z nicią
matrycowego DNA.
Najdłuższe startery mają 28-35 pz. Są stosowane w specyficznych
sytuacjach np: 1) amplifikacja blisko spokrewnionych izoform
białek; 2) amplifikacja fragmentów niekodującego DNA

Projektowanie starterów
Końce 5' i 3'
Obliczenia w biochemii i biologii molekularnej
Koniec 5' nie wymaga szczególnej uwagi, bo od niego nie zależy w
tak dużym stopniu specyficzność PCR. Przyłączać można do niego
dodatkowe nukleotydy nieparujące z matrycą np. miejsca
restrykcyjne.
Bardzo ważny dla specyficzności reakcji jest koniec 3', bo od tej
strony przyłącza się polimeraza, która zaczyna działać wtedy, kiedy
utworzy się para nukleotydów na tym końcu.
Większa zawartość par G-C w ostatnich 5 nukleotydach 3'-końca
wzmacnia siłę specyficznego wiązania startera do matrycy ale
powinno się unikać stosowania więcej niż 3 par G-C w tym
ostatnim odcinku aby nie zaszło przypadkowe parowanie
nukleotydów co obniża specyficzność reakcji.

Projektowanie starterów
Końce 5' i 3'
Obliczenia w biochemii i biologii molekularnej
Koniec 5' nie wymaga szczególnej uwagi, bo od niego nie zależy w
tak dużym stopniu specyficzność PCR. Przyłączać można do niego
dodatkowe nukleotydy nieparujące z matrycą np. miejsca
restrykcyjne.
Bardzo ważny dla specyficzności reakcji jest koniec 3', bo od tej
strony przyłącza się polimeraza, która zaczyna działać wtedy, kiedy
utworzy się para nukleotydów na tym końcu.
Większa zawartość par G-C w ostatnich 5 nukleotydach 3'-końca
wzmacnia siłę specyficznego wiązania startera do matrycy ale
powinno się unikać stosowania więcej niż 3 par G-C w tym
ostatnim odcinku aby nie zaszło przypadkowe parowanie
nukleotydów co obniża specyficzność reakcji.

Projektowanie starterów
Zawartość par G-C i ich położenie
Obliczenia w biochemii i biologii molekularnej
Par G-C powinno być w granicach 40-60% bo to daje optymalne
warunki (głównie temperaturowe) dla parowania startera z matrycą.
Należy unikać występowania więcej niż 3 par G-C obok siebie
szczególnie na 3'-końcu.
Starter powinien być bardziej „lepki” (więcej par G-C) w środku i
na 5'-końcu niż na końcu 3'.

Obliczenia w biochemii i biologii molekularnej
Projektowanie starterów
Struktury drugorzędowe
Struktury drugorzędowe starterów pojawiają się w wyniku
oddziaływań wewnątrz, bądź międzycząsteczkowych.
Powodują:
1) zmniejszenie ilości produktu PCR aż do jego
całkowitego braku;
2) hamują stapianie się starterów z matrycą;
3) w dużym stopniu zmniejszają zdolność starterów do
reakcji

Obliczenia w biochemii i biologii molekularnej
Projektowanie starterów
Struktury drugorzędowe
Self Dimer: tworzy się przez międzycząsteczkowe oddziaływanie
z drugim, takim samym starterem. Kiedy startery tworzą dimery
szybciej niż hybrydyzują z matrycą to spada wydajność PCR.
Wartości ΔG akceptowalne dla tego typu dimeryzacji: 1) dla 3'-
końca ΔG -5 kcal/mol; 2) dla środka startera ΔG -6 kcal/mol.
Przykłady
Starter ma T
m =
50°C, więc 3’ GC
homologia (ΔG=-3.1 kcal/mol) może
być problemem; jeżeli T
m
byłaby
wyższa (np. 60°C), wtedy starter
byłby dobry.
ΔG=-9.3 kcal/mol. Bardzo zły!!!
ΔG=-1.6 kcal/mol. Dobry!!

Obliczenia w biochemii i biologii molekularnej
Projektowanie starterów
Struktury drugorzędowe
Cross
Dimer:
tworzy
się
przez
międzycząsteczkowe
oddziaływanie z drugim, innym starterem kiedy występują między
nimi homologiczne fragmenty. Wartości ΔG akceptowalne dla
tego typu dimeryzacji: 1) dla 3'-końca ΔG -5 kcal/mol; 2) dla
środka startera ΔG -6 kcal/mol.
Przykłady
ΔG=-5 kcal/mol, dobry ale nie
najlepszy.
ΔG=-5 kcal/mol, dobry ale nie
najlepszy.
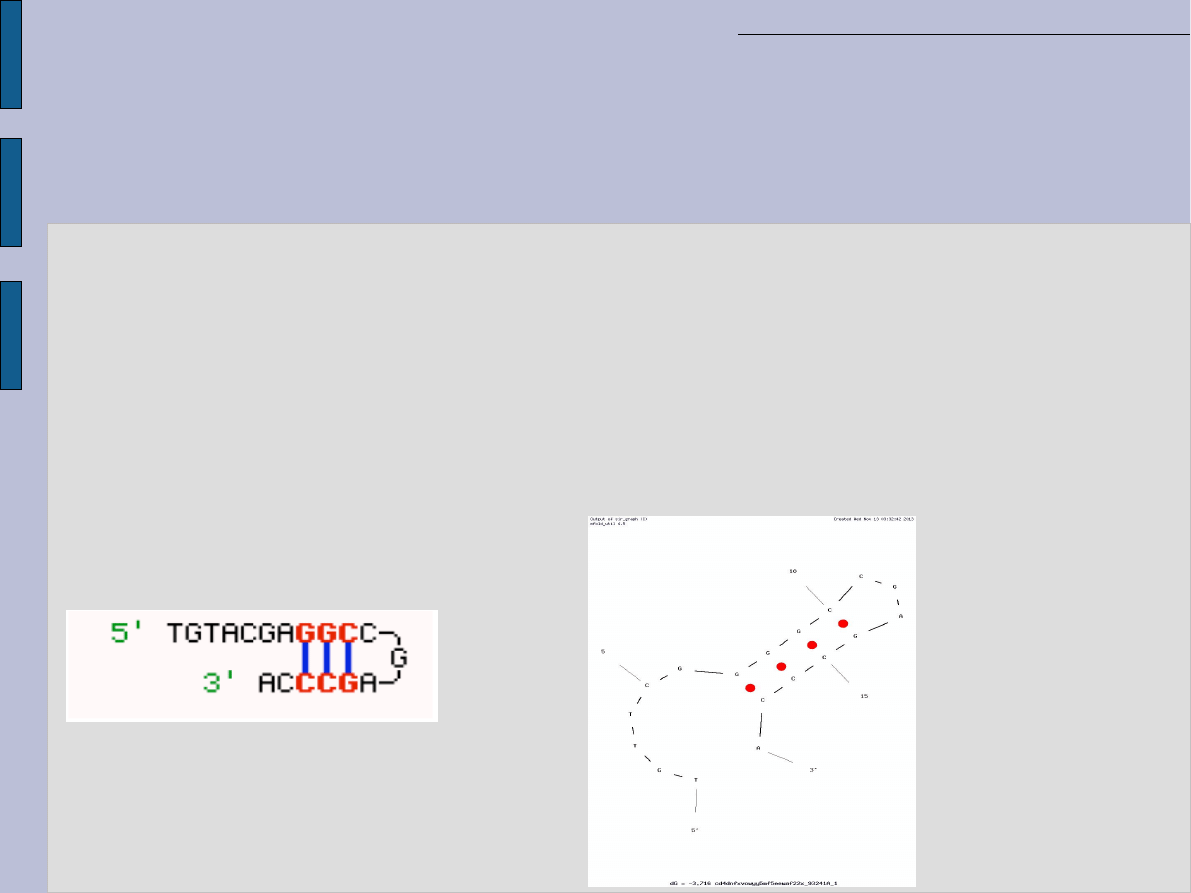
Obliczenia w biochemii i biologii molekularnej
Projektowanie starterów
Struktury drugorzędowe
Spinki (Hairpins): Tworzą się przez oddziaływania
wewnątrzcząsteczkowe w jednym starterze. Powinno się ich
unikać!!! Ale jeżeli już są to wartości ΔG akceptowalne dla tego
typu struktur: 1) dla 3'-końca ΔG -2 kcal/mol; 2) dla środka
startera ΔG -3 kcal/mol. Nie powinno być więcej niż 3 pz
wewnątrz startera, które mogą tworzyć dupleksy.
Przykłady:
ΔG=-1,36 kcal/mol, dobry ale nie
najlepszy.
ΔG=-3,72 kcal/mol,
Zły!!!

Projektowanie starterów
Powtórzenia par zasad
Obliczenia w biochemii i biologii molekularnej
Dinukleotydy: powtarzające się kolejno pary takich samych
nukleotydów (np. ATATATAT) mogą powodować nieprawidłowe
parowanie. Maksymalna, dopuszczalna ilość powtórzeń dinukleotydów
w starterze to 4.
Mononukleotydy: powtarzające się kolejno takie same nukleotydy (np.
GGGGG) mogą powodować nieprawidłowe parowanie. Maksymalna,
dopuszczalna ilość powtórzeń tej samej zasady w starterze to 4.
Wyszukiwarka
Podobne podstrony:
prezentacja do wykladu obliczenia1
prezentacja do wykladu obliczenia2
prezentacja do wykladu obliczenia1
Prezentacja do wykładu
Prezentacja do wykładu
prezentacja do wykładu pt Antropologia jako nauka
Prezentacja do wykładu systemy ogrzewania
prezentacje do wykladu WSAP 2006zaoczne
prezentacja do wykładu pt Antropologia jako nauka
Prezentacja do wykładu miejskie systemy ciepłownicze
Prezentacja do wykładu ogrzewanie powietrzem
Prezentacja do wykladu 3 2 cel
Prezentacja do wykładu
Prezentacja do wykladu 13 2 cel
Prezentacja do wykladu 14 2 cel
więcej podobnych podstron