
BIOLOGIA KOMÓRKI: 9-13 STYCZNIA 2012
Podstawy analizy strukturalnej i funkcjonalnej białek
W każdej żywej komórce białka są podstawowymi jednostkami budulcowymi i funkcjonalnymi. Począwszy od ich
roli w mechanizmach replikacji i transkrypcji DNA, a skończywszy na produkcji, przetwarzaniu i sekrecji białek,
kontrolują one procesy podziału komórek, metabolizmu, przepływ materiału i informacji z i do komórek.
Zrozumienie jak działają komórki wymaga m.inn. zrozumienia jak funkcjonują białka w komórkach, a co za tym
idzie odpowiedzi na szereg pytań takich jak lokalizacja danego funkcjonalnego białka w komórce, identyfikacja
białek i innych makrocząsteczek z którymi oddziałuje (np białkowe czynniki transkrypcyjne oddziaływać będą nie
tylko z sobą ale i z DNA), następnie określenie w jakich przedziałach komórkowych jest aktywne, jakie drogi
sygnalizacji wewnątrzkomórkowej reguluje, jaka jest jego rola we wzroście i rozwoju organizmu itd.
PRZEWIDYWANIE STRUKTURY BIAŁEK:
Z sekwencji aminokwasów: Umożliwia przewidzieć strukturę 3D i funkcję białka w oparciu o porównanie
sekwencji aminokwasów (struktury I-rzędowej) danego białka do homologicznych białek. Porównanie
umożliwiają programy dostępne nieodpłatnie w Internecie jak np BLAST (zob. rysunek poniżej). Skład
aminokwasowy
białka
można
ustalić
przez
hydrolizę
wszystkich
wiązań
peptydowych
kwasem (6M HCl, 110°C,
24h)
i
rozdział
chromatograficzny
uzyskanych aminokwasów.
Wówczas określić można
ile w danym białku znajduje
się
glicyn,
seryn
itp.
Ustalenie jednak sekwencji
aminokwasów
wymaga
podzielenia
białka
na
pewną liczbę krótszych
peptydów. W tym celu
stosuje
się
specjalne
enzymy proteolityczne (np.
trypsyna, proteaza V8) lub odczynniki (np. bromek cyjanogenu), które niszczą tylko określone wiązania
peptydowe. Każdy z peptydów jest następnie poddany degradacji Edmana w automatycznym sekwenatorze
białek. W procesie degradacji Edmana bierze udział N-końcowy aminokwas, który po potraktowaniu kwasem
jest uwalniany jako odpowiednia pochodna. Identyfikuje się ją chromatograficznie i porównuje do odpowiednich
standardów. Cykl opisanych wyżej reakcji powatrza się, by zidentyfikować kolejny aminokwas na końcu N
białka. Kolejność zaś peptydów w oryginalnym białku
ustala się poprzez sekwencjonowanie peptydów po
trawieniu białka proteazami o różnej specyficzności i
wyszukiwaniu nakładających się sekwencji aminokwasów
(zob. rysunek obok; T: trawienie trypsyną, V: trawienie
proteazą V8). Metoda ta jest jednak bardzo kosztowna i
pracochłonna, w dodatku bardzo zgrubna i nie dokladna
jeśli chodzi o przewidywanie struktury 3D białek. Dlatego
też obecnie większość sekwencji aminokwasowych białka
poznaje się obecnie w sposób pośredni poprzez
sekwencjonowanie DNA genu lub komplementarnego
DNA (cDNA).
BIOLOGIA KOMÓRKI: 9-13 STYCZNIA 2012
Podstawy analizy strukturalnej i funkcjonalnej białek
Krystalografia rentgenowska:
Podstawowa metoda
pozwalająca
z
duża
dokładnością
określić
strukturę 3D białek. Warunkiem i
najtrudniejszym etapem tej metody
jest
otrzymanie
odpowiednich
kryształów białek, które posłużą do
badań krystalografii. Promienie X
oddziałują z elektronami substancji,
przez która przechodzą. Mierząc
wzór ugięcia (dyfrakcji) strumienia
promieni X przechodzących przez
kryształ, oblicza się i ustala pozycję
atomów w krysztale. W ten sam
sposób
bada
się
kompleksy
makrocząsteczek. W kompleksach, np. enzymu z substratem, można zidentyfikować np. międzycząsteczkowe
oddziaływania odpowiedzialne za wiązanie i katalizę. Wiele białek, zwłaszcza błonowych, jest bardzo trudno
skrystalizować. Jednym z trudniejszych zadań ostatnich lat (2001) było skrystalizowanie rybosomu i
opracowanie jego struktury 3D.
Spektroskopia jądrowego rezonansu magnetycznego (NMR): Technika ta pozwala na zmierzenie relaksacji
w silnym polu magnetycznym protonów po ich wzbudzeniu falami o częstotliwości radiowej. Właściwości
relaksacji zależą od względnej pozycji protonów w cząsteczce. Badania dla białek muszą byc wielowymiarowe,
by zinterpretować nakładające się dane uzyskane z dużej liczby protonów. Dodatkowo, by polepszyć
rozdzielczość uzyskanych danych, można zastąpić w białku izotopy
12
C i
14
N odpowiednio
14
C i
15
N. Przez wiele
lat technika ta była wykorzystywana do analizy struktury małych cząsteczek. Coraz częściej technika ta jest
stosowana do analizy struktury makrocząsteczek (głównie białek) o masie do 20 kDa, a ostatnio nawet do 100
kDa i analizy poszczególnych domen białkowych. Ponadto, metoda ta nie wymaga uprzedniego skrystalizowania
próbki a jedynie odpowiednio wysokich stężeń próbki białka. Ze względu na obecność białka w roztworze można
tez badać zmiany konformacyjne białek np. w oddziaływaniach z innymi białkami, zmiany
konformacji
poszczególnych
domen
białkowych
, a także strukturę innych makroczęsteczek niż białka, jak RNA czy np.
łańcuchów węglowodanowych glikoprotein.
NOWOCZESNE METODY DYNAMICZNEGO BADANIA BIAŁEK:
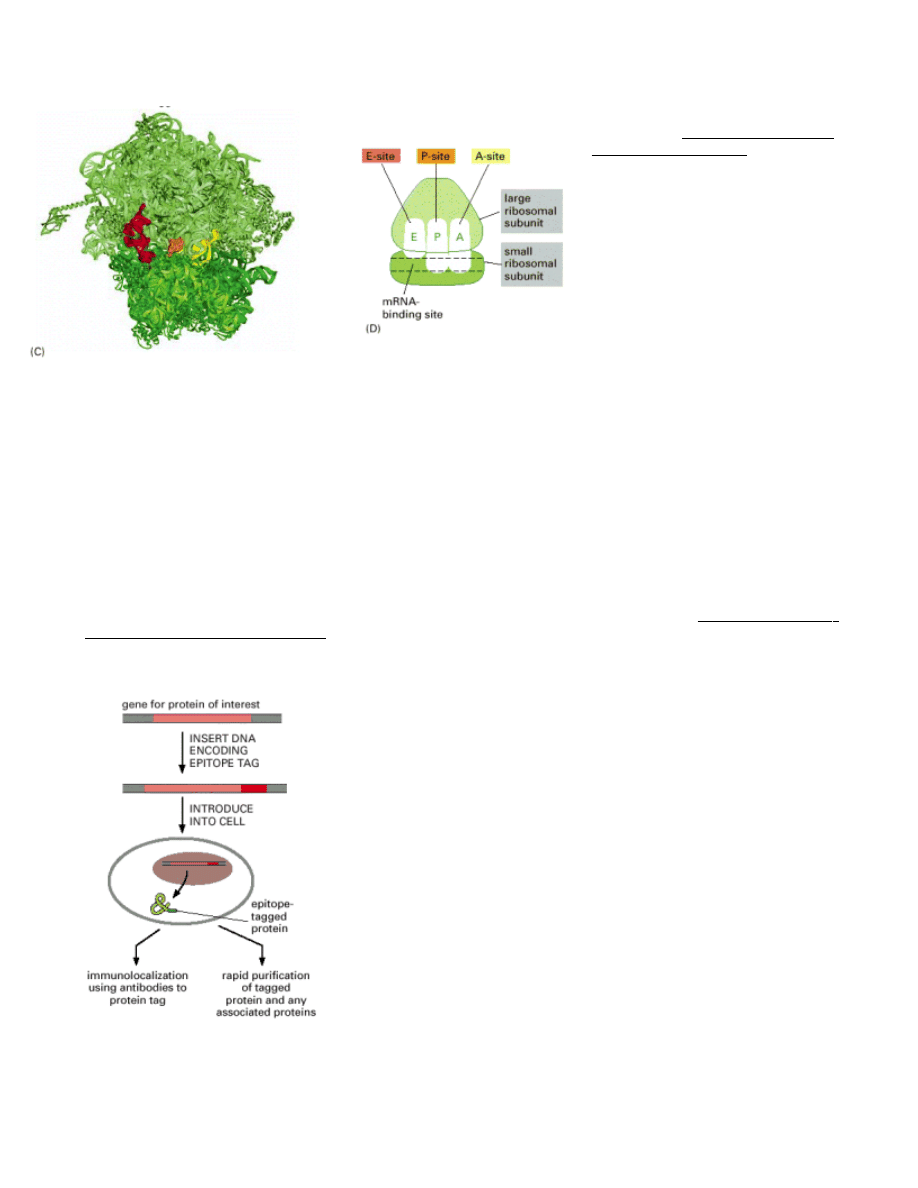
Śledzenie lokalizacji i przemieszczania białka w komórce:
Fuzja białek z GFP (z ang. GFP-Fusion proteins; GFP-Green
Fluorescent Protein) umożliwia śledzenie lokalizacji określonego białka w
komórce a tym samym przewidywanie jego funkcji w oparciu o lokalizację.
Np białko, które pod wpływem czynnika wzrostowego wędruje z
cytoplazmy do jądra może odgrywać rolę w regulacji ekspresji genów.
Wprowadzanie etykiet (z ang. epitope tagging) polega na wprowadzaniu
krótkich sekwencji kodujących kilka do kilkunastu aminokwasów.
Sekwencje te są rozpoznawane przez komercyjnie dostępne przeciwciała.
Stosując takie przeciwciała wyznakowane odpowiednim znacznikiem (np.
fluorescencyjnym) możemy podobnie jak w poprzednim przypadku śledzić
drogę białka w komórce.
BIOLOGIA KOMÓRKI: 9-13 STYCZNIA 2012
Podstawy analizy strukturalnej i funkcjonalnej białek
Poznanie partnerów z którymi dane białko oddziałuje:
Jeśli słabo scharakteryzowane białko wiąże się z białkiem o zdefiniowanych funkcjach w komórce to jest wysoce
prawdopodobne, że badane białko spełniać będzie podobne funkcje.
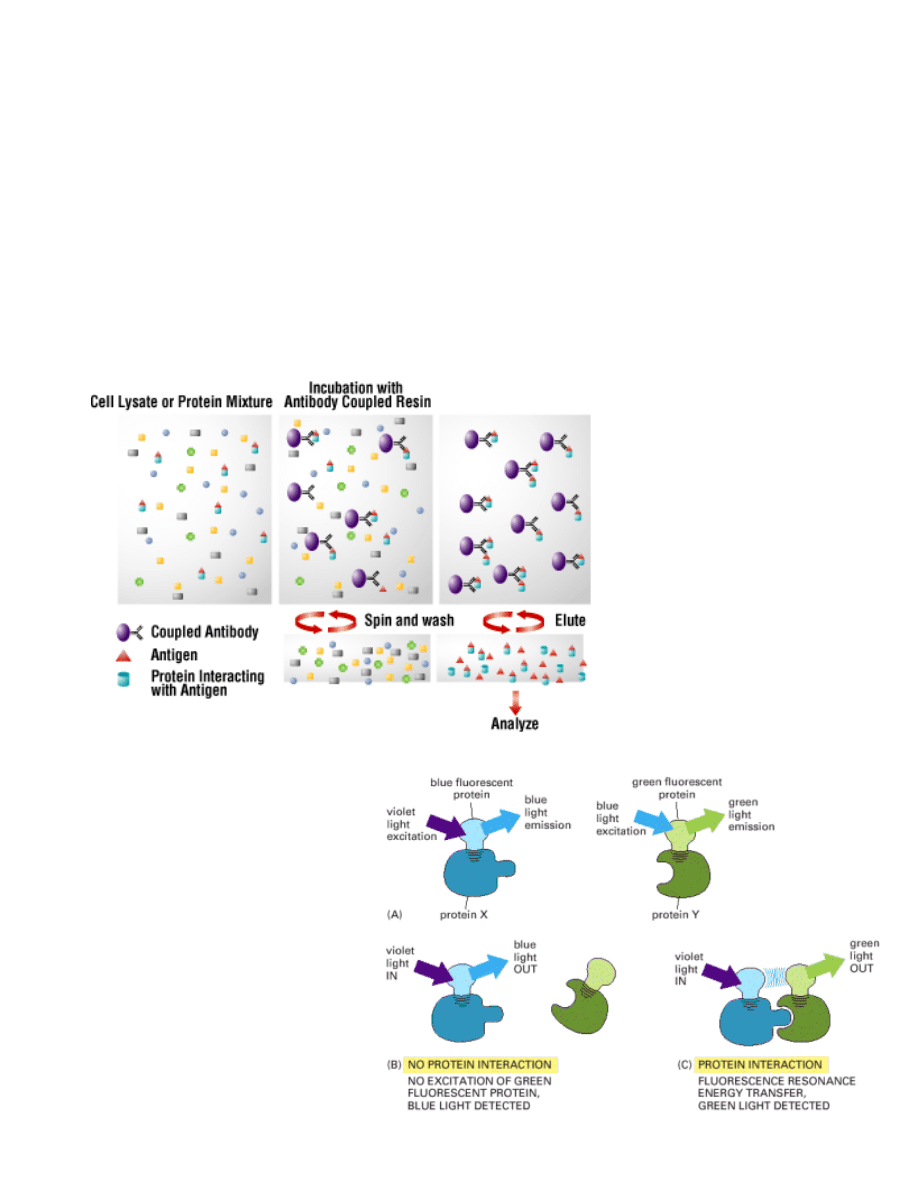
Jedną z najprostrzych metod pozwalających zidentyfikować białka wiążące się ściśle ze sobą to metoda ko-
immunoprecypitacji. W metodzie immunoprecytpitacji wykorzystujemy przeciwciała wiążące się specyficznie z
określonym białkiem, które chcemy „wyłowić” z mieszaniny różnych białek. Przeciwciała te zwykle wiąże się na
odpowiednim materiale (np. kulkach sefarozowych) umożliwiającym odseparowanie interesującego nas białka
od pozostałych (np. poprzez zwirowanie zawieszonych w mieszaninie w.w. kulek sefarozowych opłaszczonych
przeciwciałami i danym białkiem). Jeśli interesujące nas białko wiąże się ściśle z innym partnerem-białkiem, na
dnie probówki znajdzie się cały kompleks (ko-immunoprecypitacja). Takie sprecypitowane kompleksy można
następnie rozdzielić elektroforetycznie, aby np. poznać masę cząsteczkową partnerów i/lub zidentyfikować
poszczególnych partnerów z użyciem specyficznych przeciwciał dla domniemanych partnerów.
Badania odziaływania białko-białko z
wykorzystaniem
GFP
i
jego
pochodnych: Każde z interesujących
nas
białek
jest
wyznakowane
odpowiednim
znacznikiem
fluorescencyjnym (fluorochromem) w taki
sposób, że fala emitowana przez jeden
fluorochrom może być absorbowana
przez drugi. Jeśli dwa interesujące nas
białka oddziałują ze sobą (w praktyce:
zbliżają się do siebie na odległość ok 1-
10 nm), wówczas dochodzi do procesu
przekazania
energii
z
jednego
fluorochromu na drugi; proces ten jest
zwany FRET (z ang. fluorescence
resonanse energy transfer). Pomiary
FRET dokonuje się m.in. wzbudzając
jeden fluorochrom i mierząc absorbancję
drugiego.

BIOLOGIA KOMÓRKI: 9-13 STYCZNIA 2012
Podstawy analizy strukturalnej i funkcjonalnej białek
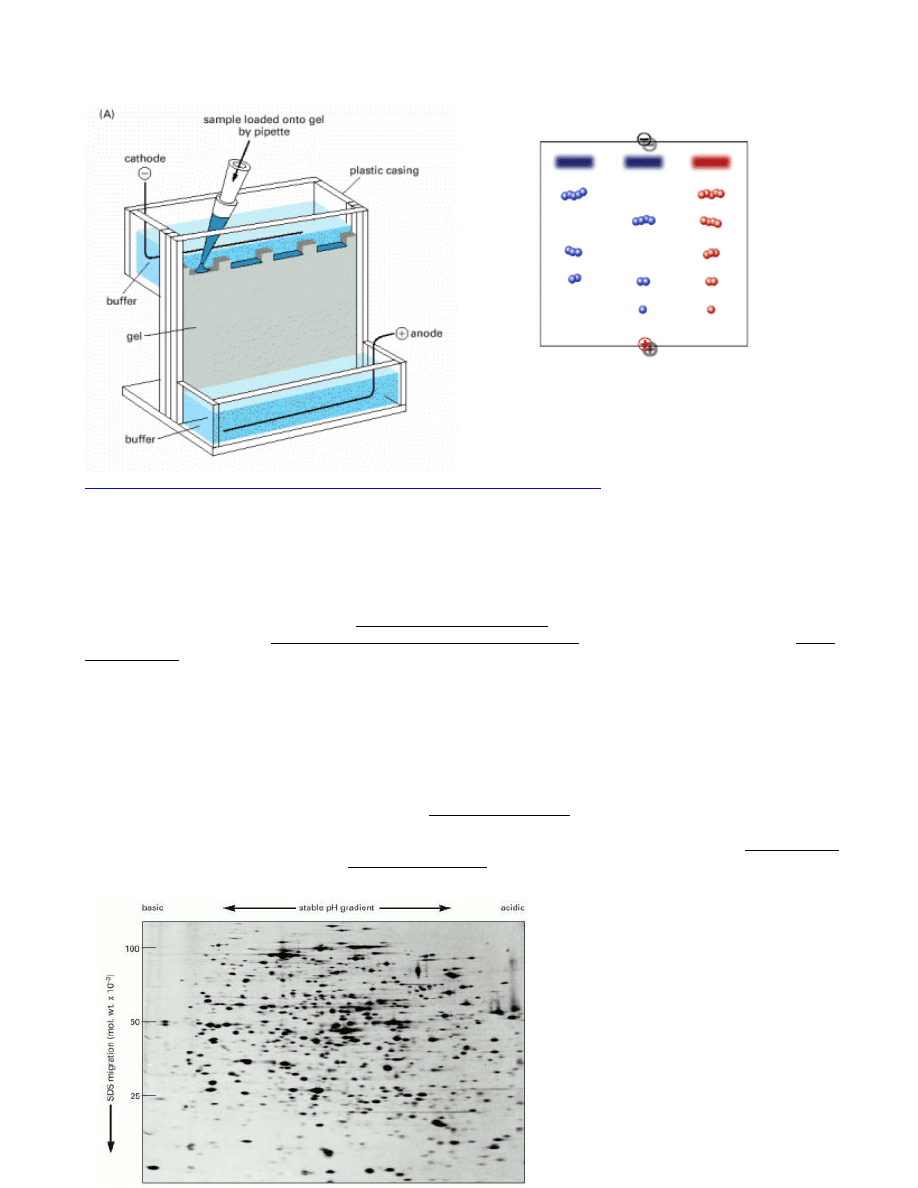
ELEKTROFOREZA BIAŁEK
Białka zwykle posiadają wypadkowy ładunek ujemny bądź dodatni, w zależności od naładowania aminokwasów,
które je budują. Kiedy
białka
są
rozdzielane
w
polu
elektrycznym,
wędrują
one
w
zależności
od
ich
wypadkowego
ładunku,
rozmiaru
oraz
kształtu
. Technika ta nazywana jest ELEKTROFOREZĄ.
Od lat 60’ych najpopularniejszym sposobem rozdziału i analizy białek jest elektroforeza w SDS na żelach
poliakrylamidowych.
Żel poliakrylamidowy jest przygotowany z monomerów tak, by pory żelu miały określoną wielkość, a białka
zawiesza się w roztworze SDS, który jest detergentem o silnym ładunku ujemnym.
Zawieszenie białek w roztworze SDS powoduje:
1.
Rozwinięcie struktury 3D białek i uzyskanie rozciągniętego
łańcucha polipeptydowego, ponieważ SDS wiąże się z rejonami
hydrofobowymi białka
2.
Dysocjację większości kompleksów białkowych i białkowo-
lipidowych do poszczególnych makrocząsteczek
3.
Opłaszczenie struktur hydrofobowych białek przez SDS i nadanie
im przeważającego ładunku ujemnego. Umożliwia to rozdział
białek w wodnych roztworach a nierozpuszczalne w wodzie białka
stają się rozpuszczalne w wodzie.
Jeśli dodamy do roztworu białka w SDS dodatkowy czynnik redukujący,
np. β-merkaptoetanol, powodujemy przerwanie wiązań dwusiarczkowych (-
S-S-) w białku, a tym samym umożliwiamy analizę poszczególnych
podjednostek polipeptydowych w makrocząsteczkach.
Rozdział białek zawieszonych w SDS na żelu poliakrylamidowym odbywa się wyłącznie
w
oparciu
o
masę
cząsteczkową danego białka ponieważ:
1.
wszystkie białka mają podobny kształt po rozwinięciu ich struktury 3D w SDS (wędrują więc z podobną
prędkością)
2.
wszystkie białka wiążą podobną ilość SDS (mają więc podobny ładunek ujemny)
Żel poliakrylamidowy działa jak sito molekularne i powoduje, że większe białka są na nim zatrzymywane dłużej,
a mniejsze wędrują szybciej.
UWAGA: Innym prostym sposobem rozdziału cząsteczek i makrocząsteczek w zależności od ich
rozmiaru jest filtracja
żelowa
(odmiana chromatografii kolumnowej).
W tej metodzie kolumna jest wypełniona porowatą macierzą i wówczas mniejsze cząsteczki zdolne
zmieścić się w porach wypełniacza zatrzymywane będą na kolumnie dłużej niż większe, które
wypłukiwane będą najszybciej z kolumny.
Po elektroforetycznym rozdziale białek, można je wybarwic bezpośrednio w żelu np. błękitem kumazyny (z ang.
Coomassie blue). Dokładniejszą detekcję zapewniają barwniki na bazie srebra i złota (do 10 ng białka).
BIOLOGIA KOMÓRKI: 9-13 STYCZNIA 2012
Podstawy analizy strukturalnej i funkcjonalnej białek
http://www.biology.arizona.edu/immunology/activities/western_blot/west1.html
ELEKTROFOREZA DWUKIERUNKOWA:
Wyspecjalizowaną odmianą elektroforezy białek jest elektroforeza dwukierunkowa, która łączy dwie metody
rozdziału białek w jedną, a mianowicie ogniskowanie
izoelektryczne
oraz elektroforezę w SDS. W pierwszym
etapie białka rozdziela się w
oparciu
o
ich
własny
ładunek
wypadkowy.
Każde białko posiada swoisty punkt
izoelektryczny, czyli takie pH, przy którym dane białko nie posiada żadnego ładunku i nie wędruje w polu
elektrycznym. W praktyce więc mieszaninę białek rozdziela się wpierw w gradiencie pH, gdzie białka wędrują do
pH odpowiadającego ich punktowi izoelektrycznemu i tam zostają. W drugim etapie rozdzielone wstępnie białka
poddaje się klasycznej elektroforezie w SDS.
W ten sposób można rozdzielić nawet do 2000 białek, czyli tyle, ile np. występuje w komórkach bakterii E.coli. W
dodatku metoda ta jest tak czuła, że pozwala rozdzielić dwa białka różniące się tylko jednym odmiennie
naładowanym aminokwasem.
Rozdzielone białka można następnie wykryć albo bezpośrednio
w
żelu
używając odpowiednio czułych metod
detekcji, takich jak barwienia na bazie srebra i złota, bądź poprzez znakowanie mieszaniny białek
radioizotopowo i detekcję rozdzielonych białek metodami autoradiografii. Alternatywnie, po przeniesieniu
rozdzielonych białek na odpowiednie filtry
nitrocelulozowe
(metoda Western blot), detekcję rozdzielonych
białek dokonuje się za pomocą przeciwciał znakowanych radioizotopowo, enzymatycznie lub fluorescencyjnie.

BIOLOGIA KOMÓRKI: 9-13 STYCZNIA 2012
Podstawy analizy strukturalnej i funkcjonalnej białek
IDENTYFIKACJA BIAŁEK (nieodzowna część PROTEOMIKI – nauki mającej na celu identyfikację i analizę
wszystkich białek występujących w danym typie komórek i rosnących w ściśle zdefiniowanych warunkach)
Po rozdziale mieszaniny białek metodą elektroforezy dwukierunkowej, poszczególne białka można poddać
analizie wycinając je z żelu i poddając trawieniu enzymami proteolotycznymi.
Trawienie białek: Np. Trypsyna
tnie
po
lizynie
i argininie
od
C-końca
każdego
białka
; a użycie bromku
cyjanogenu powoduje cięcie
wiązań
peptydowych
tylko
przy
metionine
.
Uzyskane fragmenty rozdziela się za pomocą chromatografii lub elektroforezy, a uzyskany wzór prążków (mapa
peptydowa) porównuje do istniejących map innych białek. W.w. metodę wykorzystano po raz pierwszy do
identyfikacji
mutacji
w
hemoglobinie
pacjentów z anemią sierpowatą; Wykazano różnicę w pojedynczym
peptydzie i jak się póżniej okazało była to różnica związana z zamianą jednego z aminokwasów na inny.
Obecnie w.w. metodę używa się najczęściej do identyfikacji modyfikacji
potranslacyjnych
białek
.
Spektroskopia masowa typu MALDI: Metoda ta pozwala precyzyjnie określić masę
cząsteczkową
białek i/lub
peptydów otrzymanych z nich w procesie trawienia enzymatycznego lub chemicznego. Podstawą działania
każdego spektrometru, bez względu na konstrukcję, jest jonizacja cząsteczek badanej substancji, co umożliwia
przyspieszenie jej w polu elektrycznym w próżni. Jonizację próbki można przeprowadzić na wiele różnych
sposobów (np. jonizacja chemiczna, gdzie jonizacja substancji następuje na skutek zderzeń z tzw. jonami
pierwotnymi występującymi w źródle jonów – najczęściej są to jony gazów obojętnych, metanu, izobutanu,
amoniaku). Istnieje też możliwość oznaczenia m/z substancji nie jonizujących się poprzez dołączenie (reakcja
chemiczna lub oddziaływanie fizyczne) podstawnika obdarzonego ładunkiem lub podlegającego jonizacji (tzw.
derywatyzacja) lub poprzez utworzenie adduktów np. z sodem lub potasem.Heterogeniczny strumień jonów
(dodatnich lub ujemnych) zostaje rozdzielony na szereg składowych, zależnie od stosunku masy do ładunku
(m/z). Uzyskuje się swoisty wzór mas cząsteczkowych peptydów badanego białka i porównuje go z bazą danych
opracowaną dla wszystkich dotychczas zidentyfikowanych białek, dla których masa wypadkowa i masa
cząstkowych peptydów zostały określone z użyciem podobnych metod.
Spektrometria masowa jest bardzo
czuła
, wymaga do badań niewielkiej
ilości
próbki
białka
i jest bardzo
dokładna (błąd pomiaru masy cząsteczkowej jest rzędu 0.000001), pozwala zmierzyć masę cząsteczkową białek
tak dużych jak 200 kD odpowiadających łańcuchowi polipeptydowemu o długości 2000 aminokwasów.
Metoda ta pozwala również określić sekwencję aminokwasów dla poszczególnych fragmentów peptydów a
także określić rodzaj modyfikacji tych peptydów, jak np. łańcuchy węglowodanowe, grupy fosforanowe, grupy
metylowe itp. Dlatego metoda ta stała się konkurencyjna w stosunku do degradacji Edmana jako metoda
sekwencjonowania białek i umożliwia identyfikację modyfikacji posttranslacyjnych, czy analizę związków
endogennych występujących w bardzo niskich stężeniach. Pojedyncze aminokwasy posiadające interesujące
nas modyfikacje mogą być wykryte, ponieważ z użyciem tej metody bedą wykazywały charakterystyczny wzrost
masy cząsteczkowej. Technika ta znajduje również zastosowanie w dziedzinach takich jak ochrona środowiska,
kontrola antydopingowa, farmakologia, diagnostyka medyczna, biotechnologia czy ostatnio w dynamicznie się
rozwijającej proteomice.
Wyszukiwarka
Podobne podstrony:
ANALIZA ANIONÓW - ćwiczenia z 13.11, ANALIZA ANIONÓW
Cwiczenia 14 Podstawy analizy RNA i DNA
Podstawy analizy 2 Cwiczenia, zadania
PODSTAWY REKREACJI CZASU WOLNEGO- ćwiczenia, GWSH, podstawy rekreacji i czasu wolnego
HARMONOGRAM ĆWICZEŃ 13 tematy 1
Fizjologia Ćwiczenia 13
podstawy analizy niepewności pomiarowych
m10 ekologia cwiczenie 13
cwiczenie 13
Ćwiczenie 13a, Patofizjologia, Ćwiczenia 13-15 (wydalniczy, nerwowy, nowotwory, toksykologia, rytmy
Ćwiczenie 13, Immunologia
Metabolizm tłuszczowców, Patofizjologia, Ćwiczenia 13-15 (wydalniczy, nerwowy, nowotwory, toksykolog
Podstawy analizy fundamentalnej Nieznany
Cwiczenie 13 o
Ankieta CWICZENIA, matura podstawowa pisemna
więcej podobnych podstron