
Niekodujące RNA
Niekodujące RNA
&
&
Terapeutyczne kwasy
Terapeutyczne kwasy
nukleinowe
nukleinowe
Barbara Licznerska, 2009
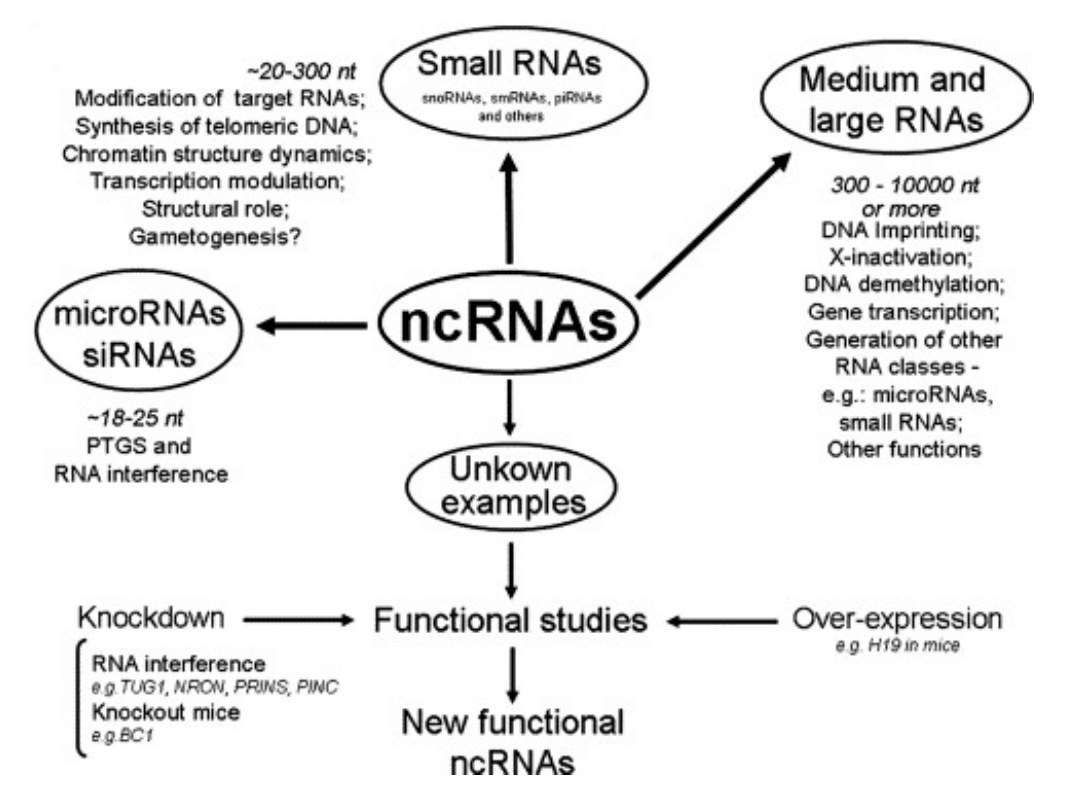
Niekodujące RNA (ncRNA)
•
tRNA
•
rRNA
•
snRNA
•
snoRNA
•
miRNA
•
siRNA (np. rasiRNA, tasiRNA)
•
piRNA
•
(rybozymy)
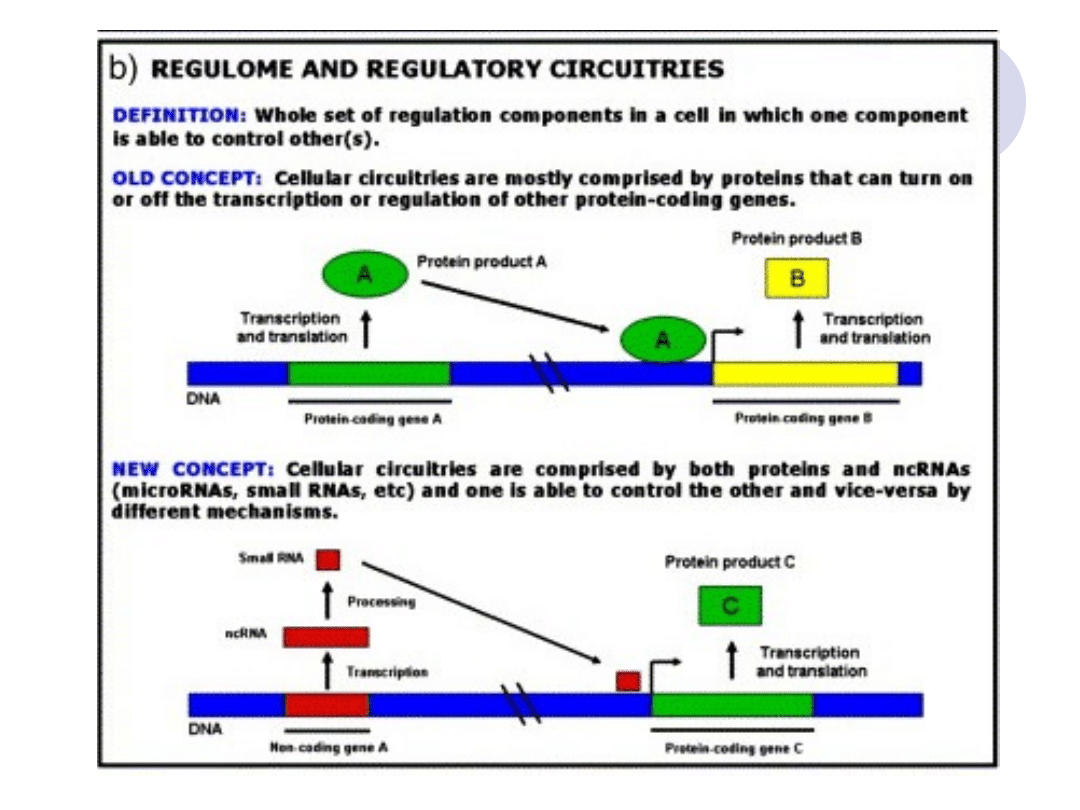
Regulacja ekspresji genów
Prokariota
-
dominują w genomie sekwencje
kodujące białka (80-95%)
-
głównie model operonu
Eukariota
-
dominują sekwencje „niebiałkowe”
-
ncRNA istotne w regulacji

1) snoRNA
small nucleolar RNA (małe jąderkowe RNA)
długość 60-300 nt
większość kodowana przez introny
dwie główne klasy:
-
box C/D snoRNA
-
box H/ACA snoRNA
rola: modyfikacje rRNA
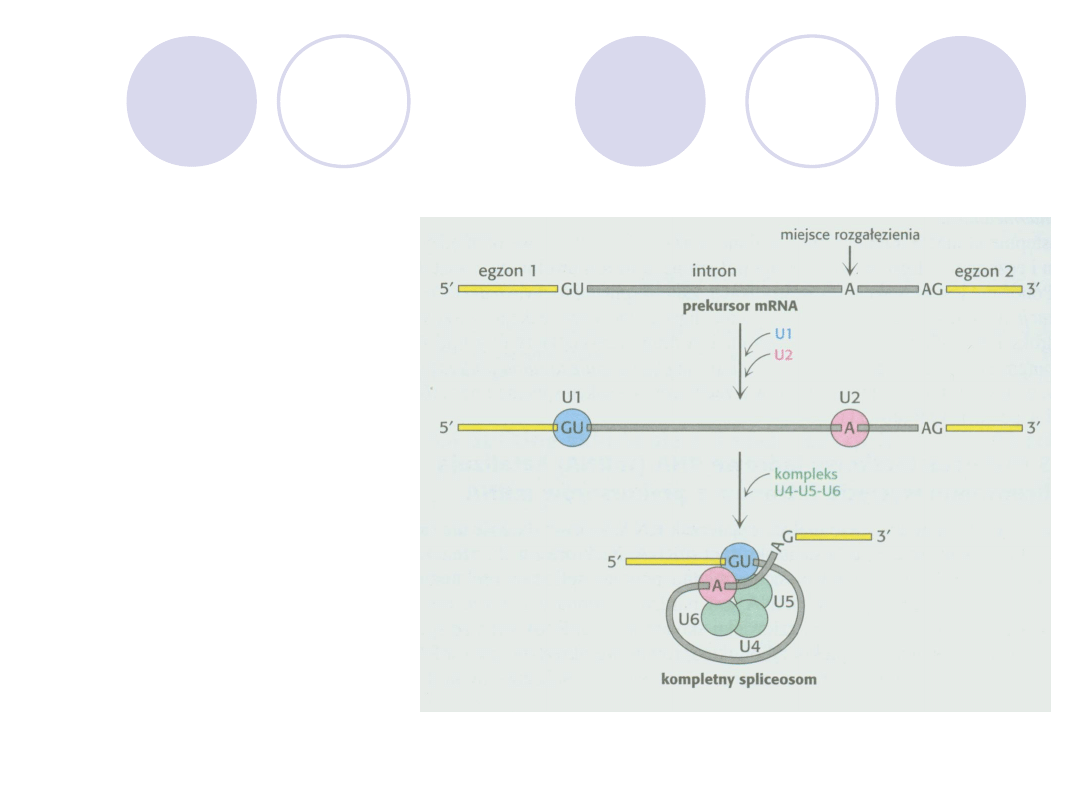
2) snRNA
small nuclear
RNAs
(małe jądrowe
RNA)
splicing

3) mikroRNA (miRNA)
1-niciowa struktura
21-23 nt
struktura niedojrzała tworzy
struktury „spinki do włosów”
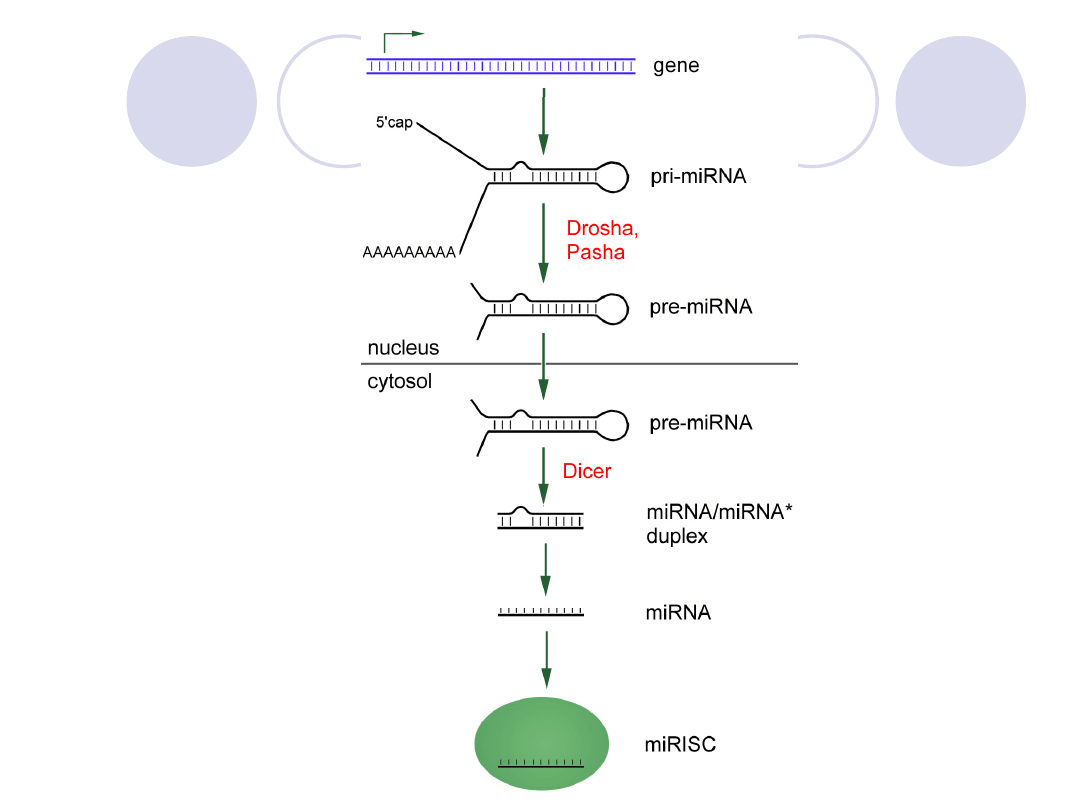

Sposób działania miRNA
1)
Regulacja ekspresji genów
a.
nukleolityczna degradacja mRNA
b.
transkrypt bez zmian, zahamowana
translacja
2)
Wyciszanie ekspresji genów
a.
degradacja transkryptu
b.
zahamowanie translacji

Rola miRNA -
przykłady
rozwój i dojrzewanie osobników
różnicowanie komórek macierzystych
segregacja chromosomów
profil ekspresji genów poszczególnych tkanek
kontrola namnażania komórek
kontrola apoptozy
udział w przekazywaniu sygnałów ?
udział w mechanizmie obrony przed wirusami
?

Zastosowanie miRNA
(przykład)
określanie profilu ekspresji miRNA
w tkance nowotworowej:
->diagnostyka
-> prognozowanie
-> leczenie nowotworów
np. miR-15 i miR-16 w przebiegu
przewlekłej białaczki limfocytowej

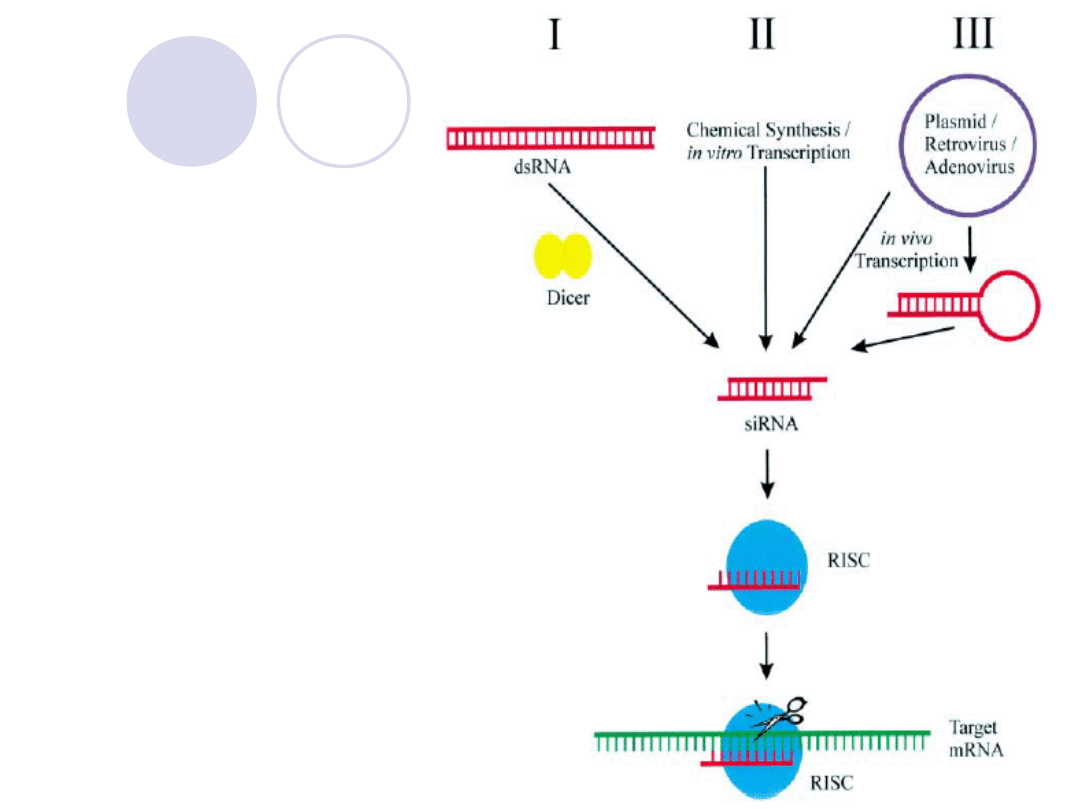
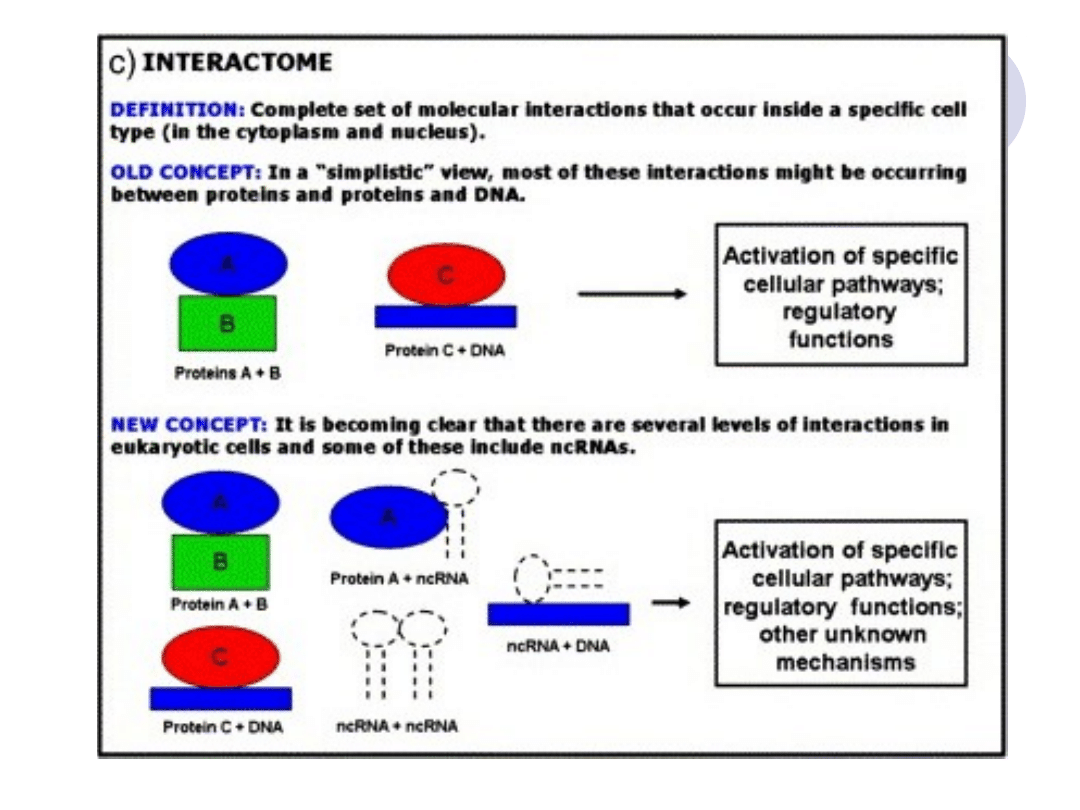
4) Interferujące RNA (siRNA)
short interfering RNA
(krótkie interferujące RNA)
długość 23-27 nt
niedojrzała postać 2-niciowa
niesparowane dwa nt na końcu 3’
brak struktury „spinki do włosów”
egzo i endogenne
rasiRNA
repeat-associated RNA
(siRNA związane z sekwencjami powtórzeniowymi)
główna klasa
siRNA
kodowane głównie przez sekwencje
heterochromatynowe obszarów
centromerowych i telomerowych
kompleks RITS zamiast RISC

tasiRNA
trans-acting RNA (siRNA działające in
trans)
długość ok. 21 nt
dot.
ROŚLIN
(jak dotychczas)
jako jedyne
siRNA
wpływają na
ekspresje innych genów niż te, z
których się wywodzą
Eur J Biochem 270, 1628, 2003
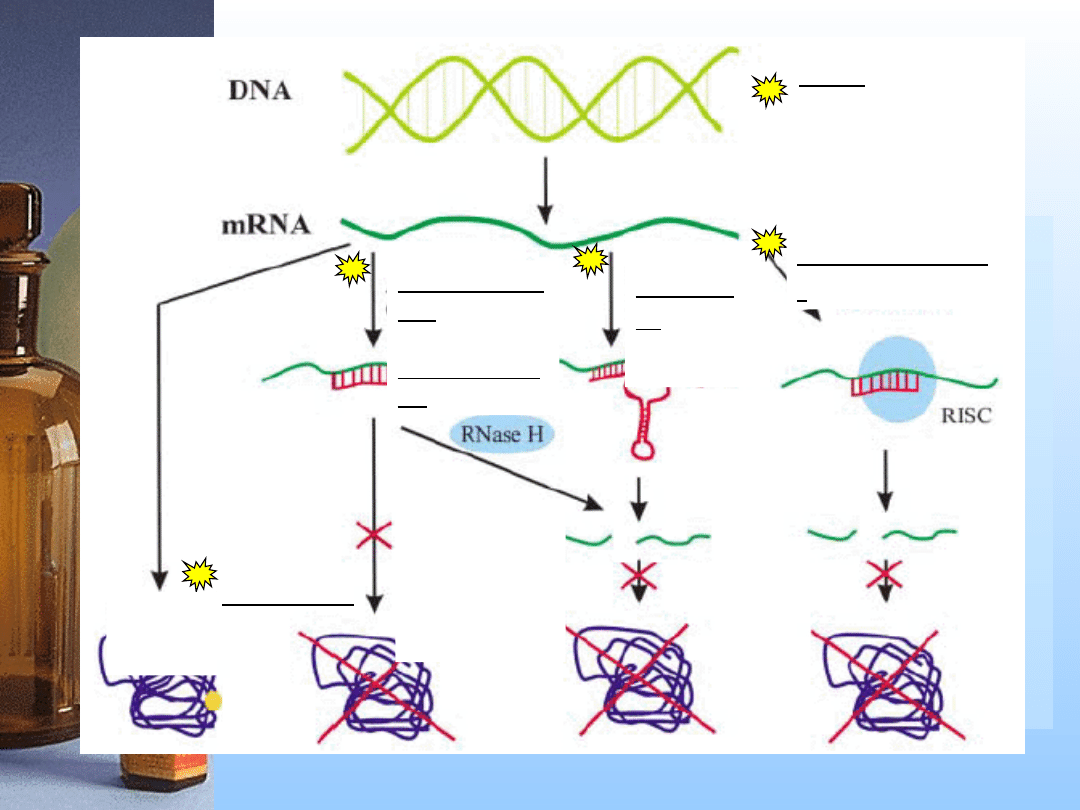
Mechanizm
działania
siRNA

Mechanizm interferencji RNA
(RNAi)
miRNA
&
siRNA

5) piRNA
PIWI-interacting RNA
długość 26-31 nt, struktura I rzęd.
przepisywane w postaci długich
transkryptów; dojrzewanie dzięki
nukleazom z rodziny RNazy III
rola …
Gene: 386 (2007) 1-10
Gene 386 (2007) 1-10
Gene 386 (2007) 1-10
Gene 386 (2007) 1-10

ANIMACJE
http://www.nature.com/nrg/journal/v2/n2/
animation/nrg0201_110a_swf_MEDIA1.ht
ml
http://www.nature.com/focus/rnai/animati
ons/index.html

INNE ŹRÓDŁA
dotychczas opisane miRNA:
znane sekwencje genów małych ncRNA:

TERAPEUTYCZNE
KWASY NUKLEINOWE
TFO
antysensowne oligonukleotydy
rybozymy
egzogenne siRNA
aptamery
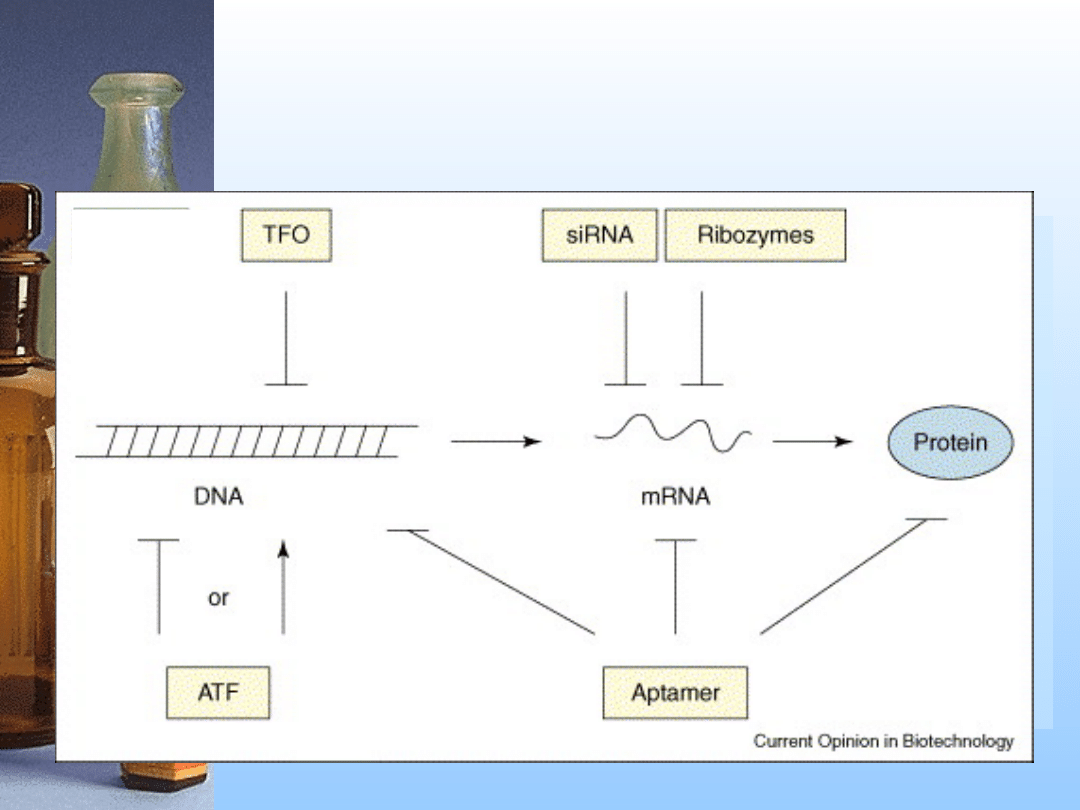
ZAHAMOWANIE EKSPRESJI GENU
I FUNCKJI BIAŁKA
Eur J Biochem 270, 1628,
2003
TFO
Aptamer
Oligonukleo
tyd
antysensow
ny
Rybozy
m
siRNA
Białk
o
lek
zahamow
.
translacj
a
1. Oligonukleotydy
ANTYGENOWE (TFO)
Jednoniciowe kwasy nukleinowe,
które blokują transkrypcję danego
białka poprzez wiązanie się z
komplementarną
dla niego sekwencją DNA
(blokują tzw. matrycę danego białka),
tworząc tzw. triplet
TFO – triplet forming oligonucleotides

2. Oligonukleotydy
ANTYSENSOWNE (ASO)
Jednoniciowe kwasy nukleinowe,
które blokują translację danego
białka poprzez wiązanie się z
komplementarną dla niego
sekwencją mRNA
Eur J Biochem 270, 1628,
2003
MECHANIZ
M
DZIAŁANIA
ASO
Przyłączenie
RNazy H
RNaza H
antysensowny oligonukloetyd
chimeryczny
Zahamowanie translacji
Rybosom
antysensowny
oligonukleotyd

Mechanizm działania ASO
inhibicja mRNA poprzez:
* hamowanie transportu mRNA z
jądra
* hamowanie splicingu
* zahamowanie translacji
poprzez:
- zawada przestrzenną
- aktywację RNazy H

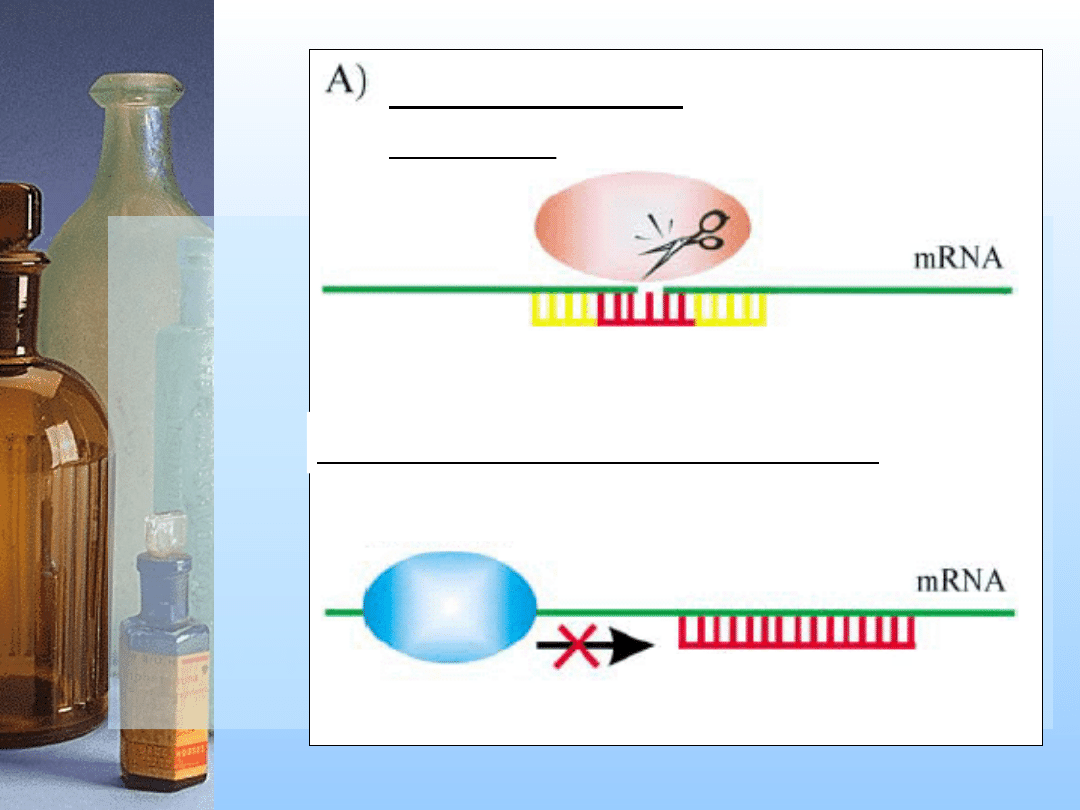
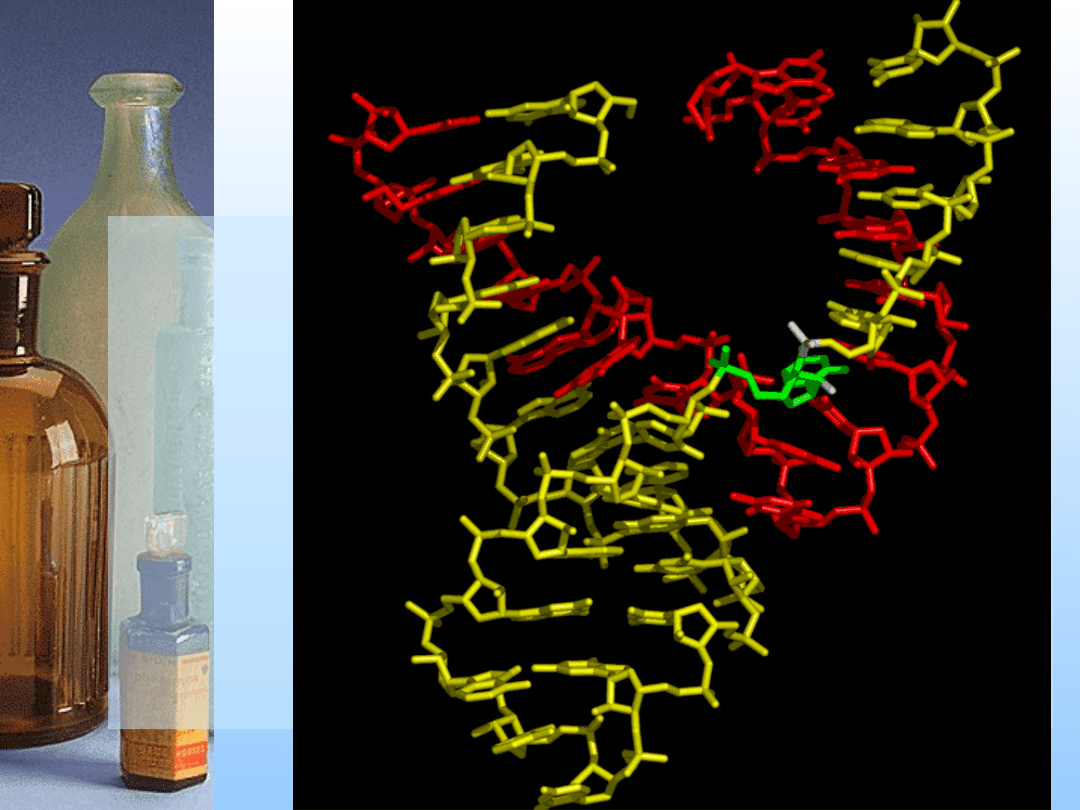
3. Rybozymy
katalityczne cząsteczki RNA
rozkładające docelowe RNA
lub
naprawiające zmutowane
komórkowe RNA
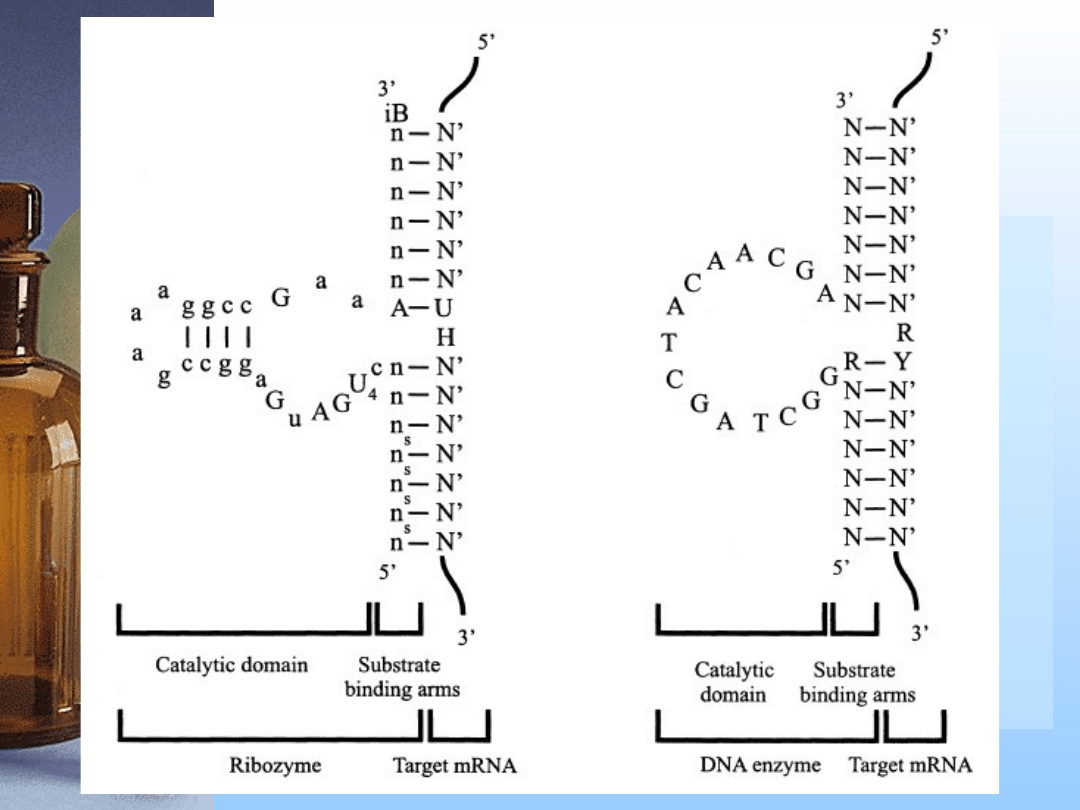
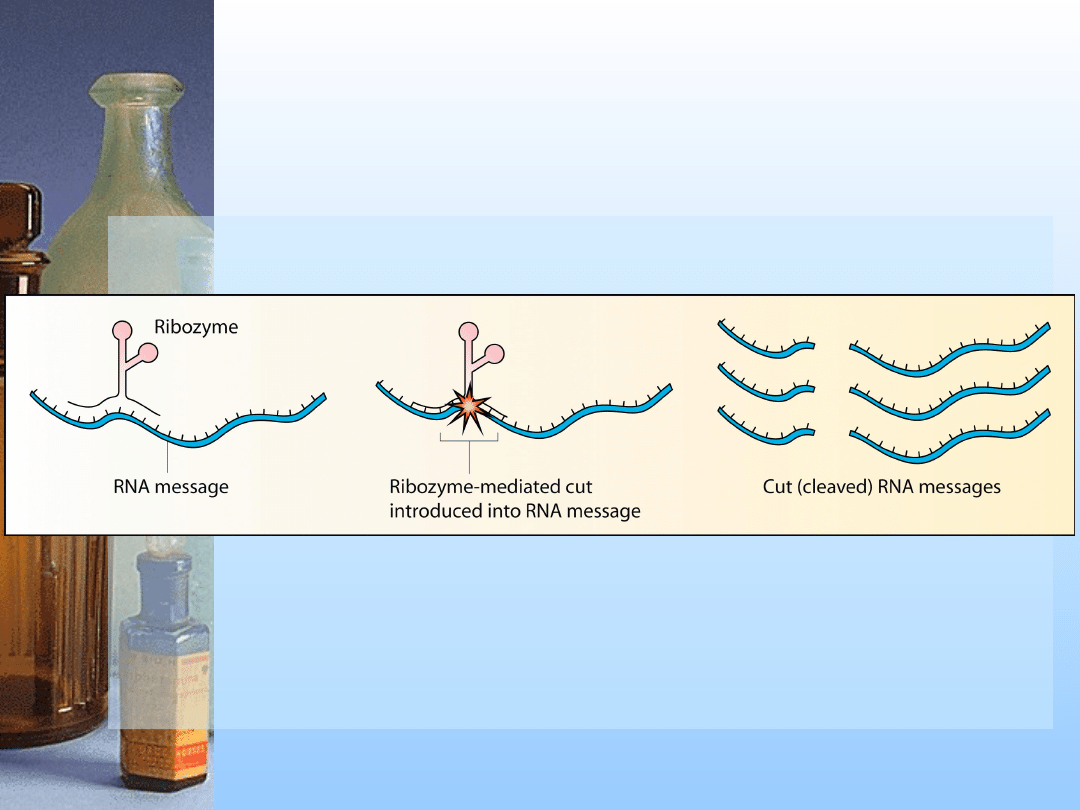
Mechanizm działania
rybozymów
DZIAŁANIE
RYBOZYM
U

Rybozymy typu
„hammerhead”
* małe, specyficzne, prosty mechanizm
rozszczepiania
* skuteczniejsze od ASO???
– wydajność
– stała inaktywacja genu
– niższe stężenia terapeutyczne
– specyficzność

4. siRNA
Dwuniciowe, krótkie kwasy
nukleinowe, powodujące
enzymatyczny rozkład mRNA, wobec
którego komplementarna jest nić
antysensowna siRNA
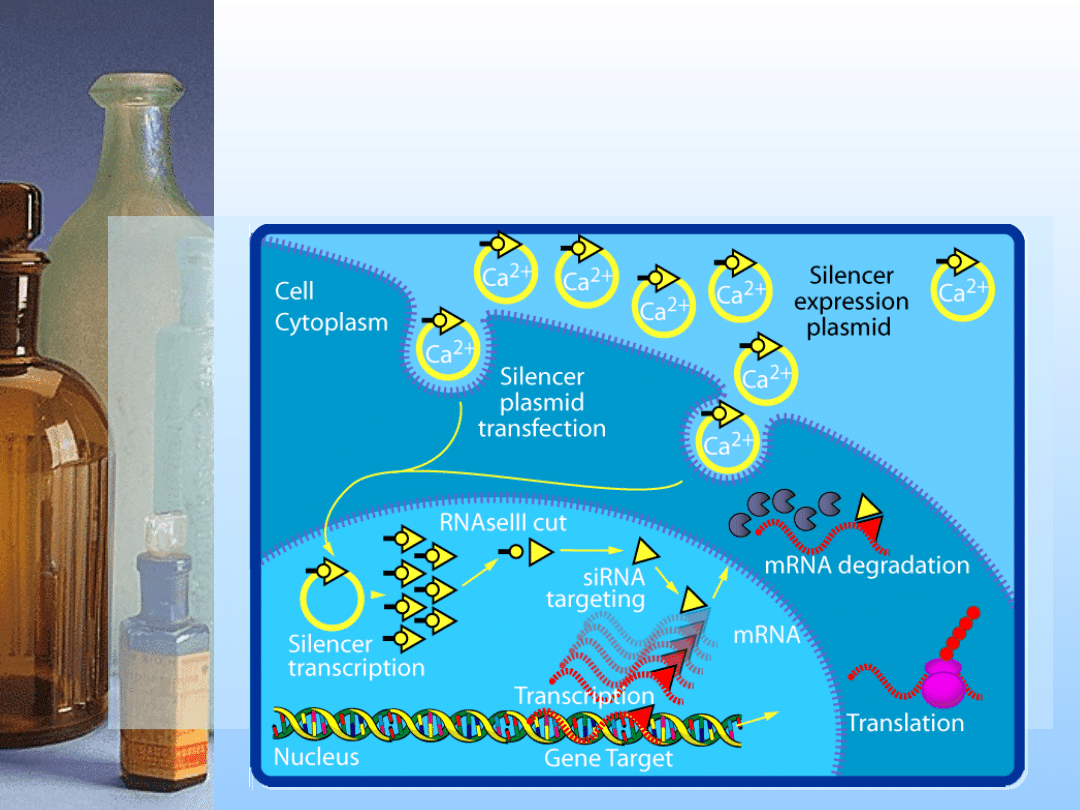
Mechanizm działania
egzogennego siRNA
tzw. potranskrypcyjne wyciszanie genów = PTGS

5. Aptamery
Jedno- lub dwu-niciowe kwasy
nukleinowe (DNA lub RNA),
których trójwymiarowa struktura
umożliwia selektywne rozpoznawanie
różnych cząsteczek, np. białek
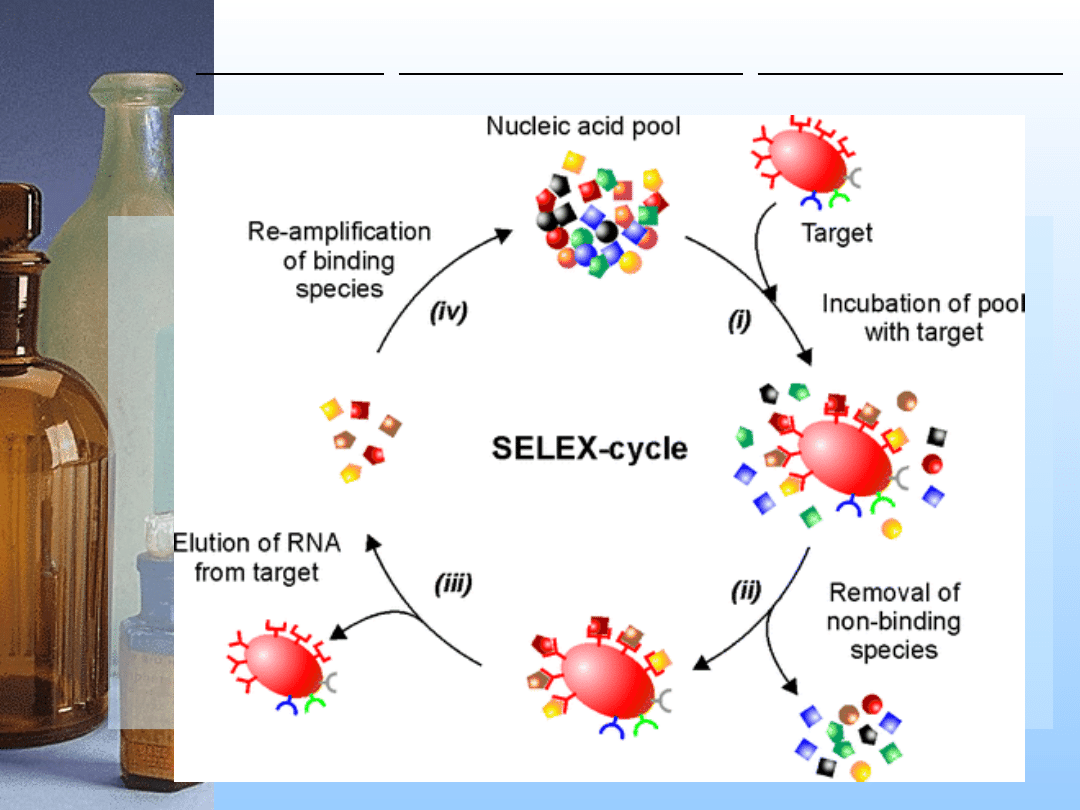
PROCES IDENTYFIKACJI APTAMERÓW
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
Wyszukiwarka
Podobne podstrony:
OBLIGACJE 10 11 2009 dla studentów
OBLIGACJE 10 11 2009 dla studentów
0 instrukcja BHP dla studentow 2009, Biologia molekularna, laborki
Nadci¶nienie prelekcja dla studentów V roku 2009 wersja dl astudentów
zestaw pytan dla studentow 2009, notatki, testy, Ekologia, fwdegzaminy
Pytania na kolokwium II sem - dla studentow 2009, uczelnia WSEI Lublin, UCZELNIA WSEI 2 1, MATERIAŁY
Kiła dla studentów 2009
Kanon lektur dla studentów filologii germańskiej w IFG UAM (nowy kanon studia dwustopniowe, wersja
Bilikiewicz, Psychiatria podręcznik dla studentów (2009), 270 307, 323 340
gruźlica dla studentów2
Prezentacja 2 analiza akcji zadania dla studentow
Kosci, kregoslup 28[1][1][1] 10 06 dla studentow
higiena dla studentów 2011 dr I Kosinska
więcej podobnych podstron