
Przebieg i regulacja
procesu translacji
Wykonały:
Monika Letkiewicz
Agnieszka Warcaba
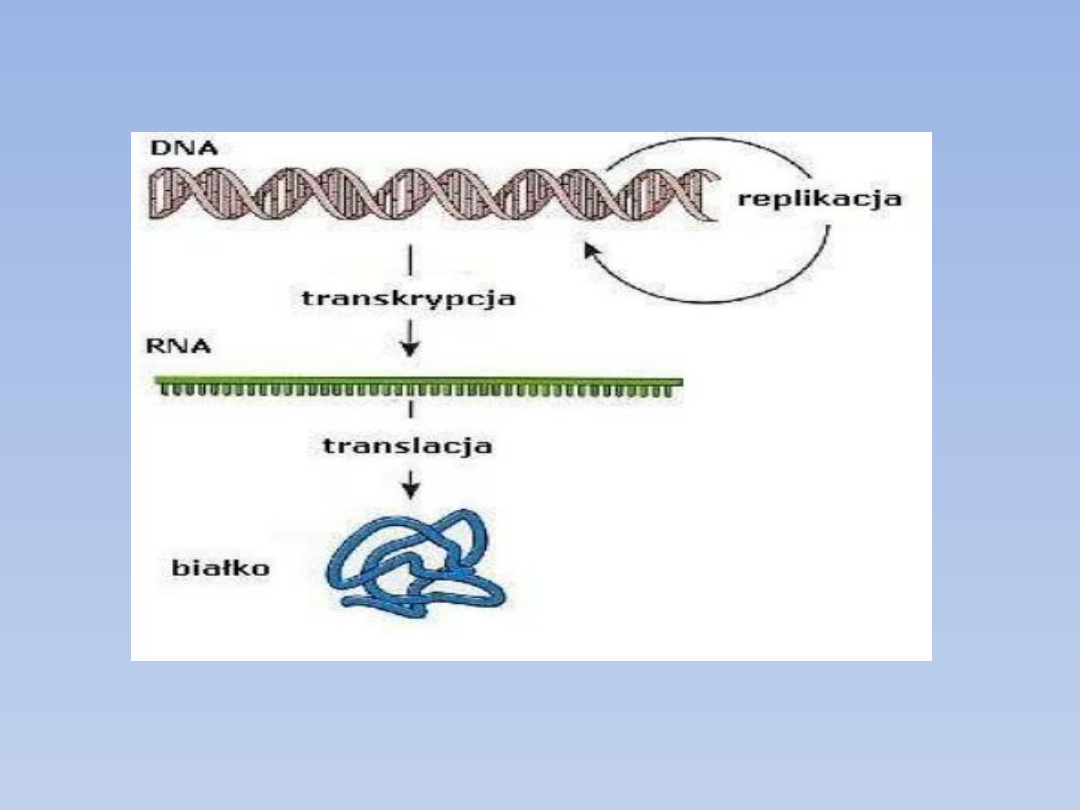
Translacja stanowi drugi etap biosyntezy biała
Uproszczony schemat przebiegu biosyntezy białka
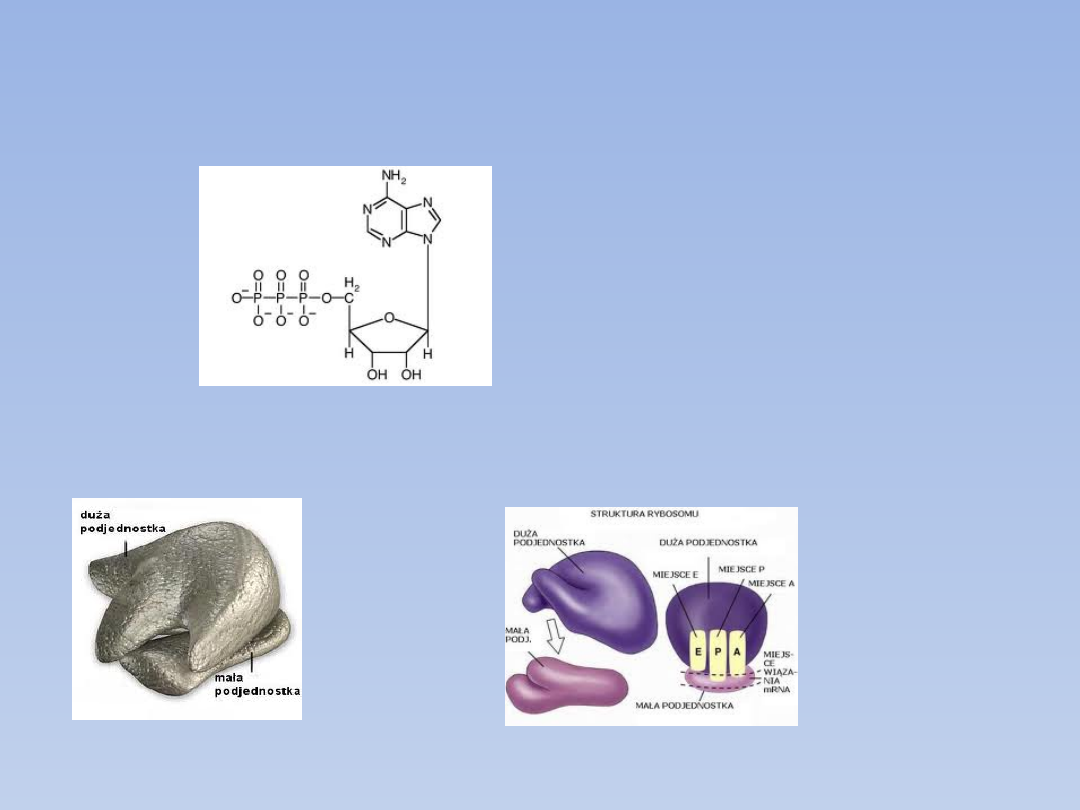
Elementy niezbędne do przebiegu translacji:
•ATP (adenozynotrifosforan)
•Rybosomy (zbudowane z 2 podjednostek)
•Aminokwasy
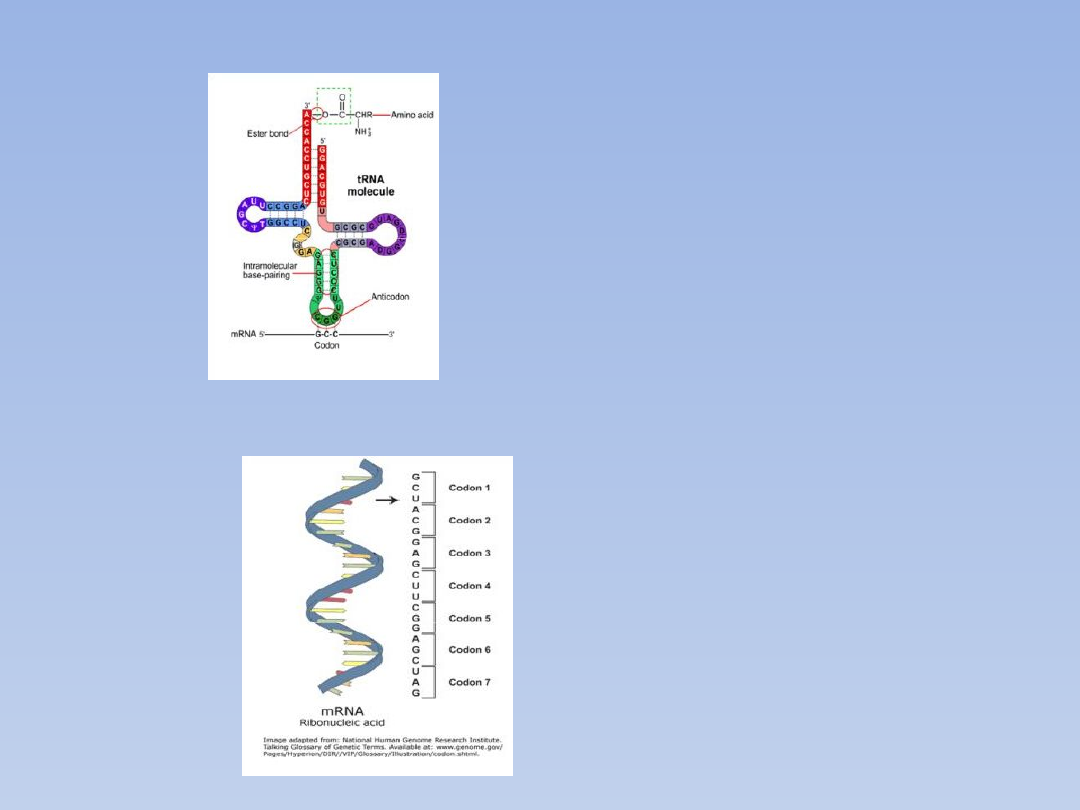
•tRNA
•mRNA

Na podstawie informacji zawartej w mRNA, w drugim etapie
ekspresji genu, syntetyzowane jest białko o określonej sekwencji
aminokwasów.
Następuje tu przetworzenie „języka kwasów nukleinowych” w
cząsteczce mRNA na „język aminokwasów” w cząsteczce białka.
Translacja mRNA, czyli biosynteza na rybosomie, jest końcowym
etapem w procesie ekspresji informacji genetycznej zawartej w
sekwencji nukleotydów w mRNA na kolejność aminokwasów białku
składa się (podobnie jak replikacja DNA i transkrypcja) z 4 faz:
AKTYWACJI
INICJACJI
ELONGACJI
TERMINACJI

Aktywacja

W aktywacji właściwy aminokwas jest dołączany do właściwego tRNA
za pomocą wiązania estrowego.
Zanim aminokwas będzie mógł przyłączyć się do tworzącego się
łańcucha polipeptydowego, musi zostać on aktywowany. W proces ten
włączany jest:
-tRNA
-syntetaza aminoacylo-tRNA
-ATP, będący źródłem energii reakcji
Proces ten przechodzi w 2 etapach, katalizowany jest przez enzym-
syntetazę aminoacylo-tRNA. W drodze tej reakcji aminokwas zostaje
aktywowany i utworzony zostaje aminoacylo-tRNA.
•Etap pierwszy:
Polega na aktywowaniu aminokwasów- czyli przeniesieniu ich na
wyższy poziom energetyczny.
Synteaza aminoacylo-tRNA aktywuje aminokwas przez przyłączenie
AMP do grupy karboksylowej aminokwasu pochodzącego z cząsteczki
ATP i odłączenie difosforanu.
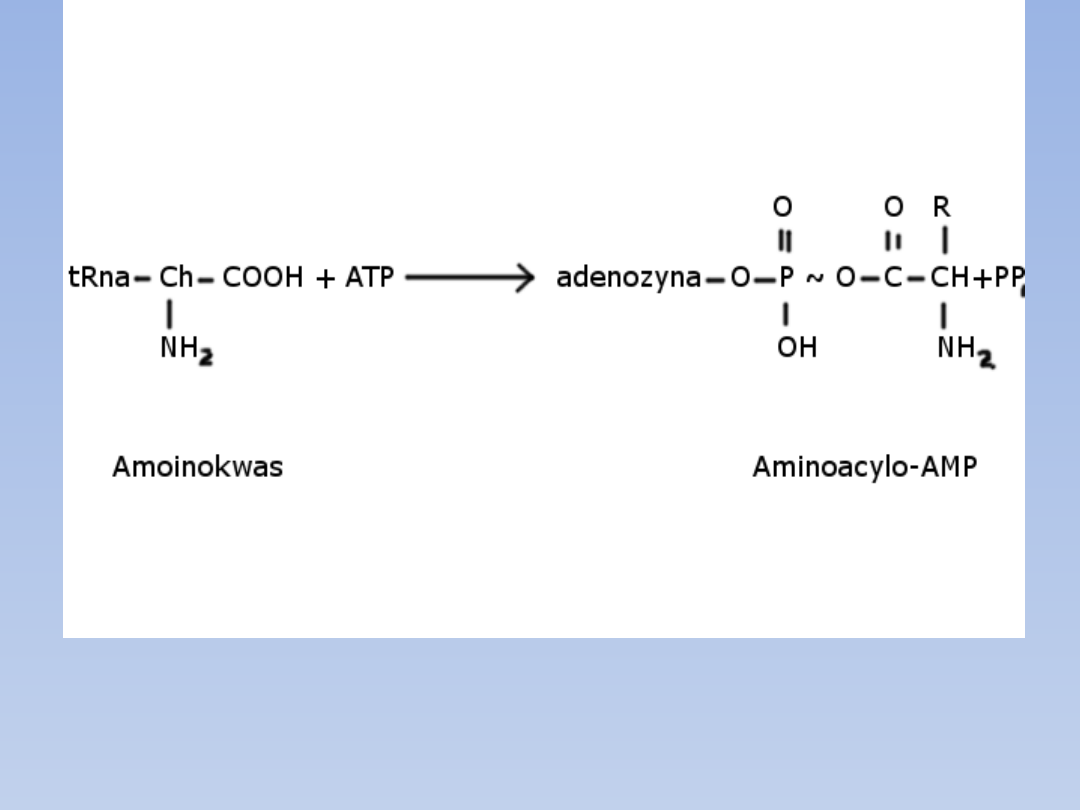
Aktywowany aminokwas pozostaje związany z cząsteczką synteazy.

•Etap drugi:
Enzym Syntetaza aminoacylo-tRNA katalizuje przeniesienie grupy
aminoacylowej (R-CH
2
NH
2
-CO-) z aminoacylo-AMP na odpowiedni
rodzaj tRNA.
Aminokwas + ATP Aminoacylo-AMP + PP
Aminoacylo-AMP + tRNA Aminoacylo-tRNA + AMP
Aminokwas + ATP + tRNA aminoacylo-tRNA + AMP + PP
Grupa aminoacylowa przyłącza się do końcowego nukleotydu
adeninowego tRNA, znajdującego się w tryplecie CCA, tj na końcu 3’
ramienia aminokwasowego (akceptorowego) tRNA wiążąc się estrowo
z 3’ grupą hydroksylową rybozy. Zaaktywowane i związane z tRNA
aminokwasy są przenoszone do formującego się polirybosomu, gdzie
ustawiają się w takiej kolejności, jak to wyznaczają sekwencje trójek
nukleotydowych (kodonów) w mRNA oraz odpowiadające im
antykodony w tRNA.

Inicjacja

Inicjacja translacji ma miejsce, kiedy mała podjednostka rybosomu
przyłącza się do końca 5' mRNA
Zapoczątkowanie formowania się łańcucha polipeptydowego polega
na wytworzeniu tzw. kompleksu inicjacyjnego, który składa się z:
-rybosomu
-mRNA
-inicjatorowego tRNA – N-formylometionylo-tRNA u organizmów
prokariotycznych lub metionylo-tRNA u organizmów eukariotycznych
-białkowych czynników inicjacyjnych- IF u eukariotów i eIF (eucaryotic
initiation factors) oraz GTP
Podczas inicjacji rybosom i inicjator tRNA muszą właściwie rozpoznać
miejsce na nici mRNA, w którym powinna się rozpocząć biosynteza
białka, co warunkuje dalszy prawidłowy odczyt.
Proces inicjacji łańcucha u eukariotów nie jest tak dobrze znany
jak w wypadku bakterii. Wiadome jest, że istnieje między nimi kilka
różnic.
-do inicjacji wykorzystywana jest metionina
-istnieje przynajmniej 8 czynników inicjacji

Proces inicjacji łańcucha u bakterii posiada kilka etapów
obejmujących:
-interakcję mRNA,
-dużej i małej podjednostki rybosomu,
-inicjatorowego tRNA,
-GTP
-3 czynników inicjacyjnych- IF1, IF2, IF3
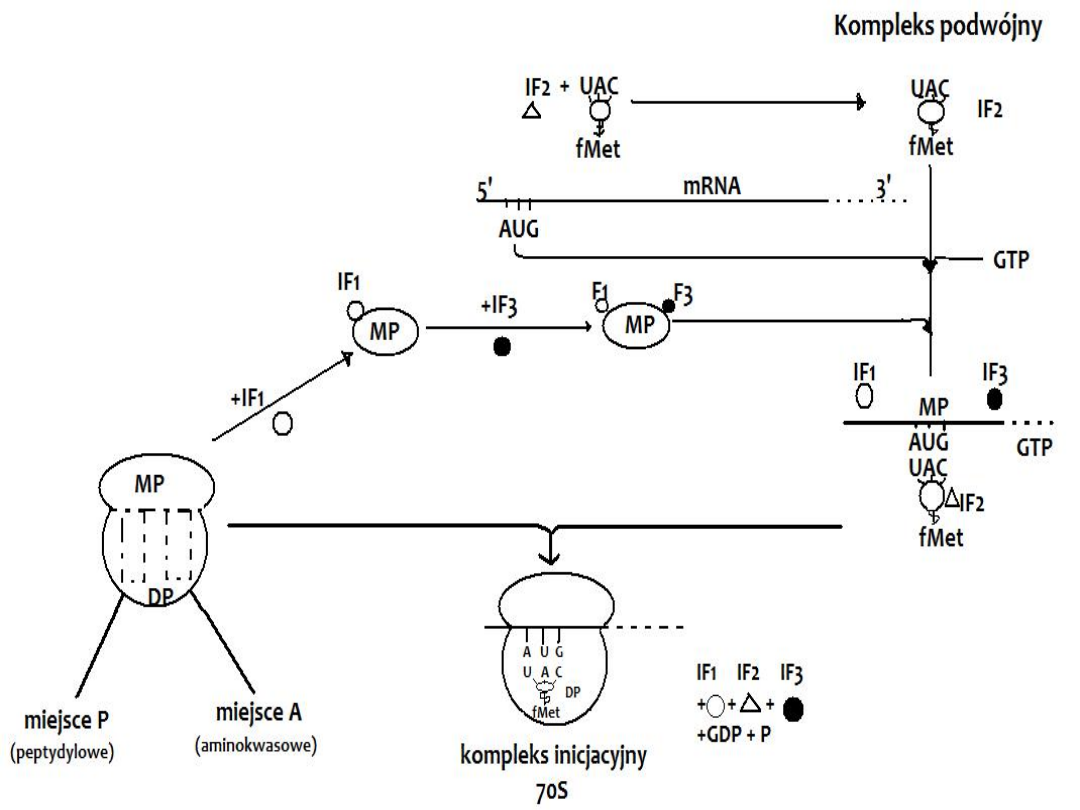
Pierwszą reakcją inicjacji jest dysocjacja rybosomu na dwie oddzielne
podjednostki- małą (MP) i dużą (DP) Dysocjacja jest stymulowana
przez czynnik inicjacji- 1 (IF1), który łączy się bezpośrednio z małą
podjednostką rybosomu. Odłączona mała podjednostka jest
stabilizowana przez przyłączenie IF3, który działa jako „czynnik
antyasocjacyjny” zapobiegający ponownemu połączeniu się małej
podjednostki z dużą
Mała podjednostka jest przygotowana do następnego etapu, w którym
mRNA i inicjatorowi tRNA w połączeniu z aminokwasem (fMEt) są
przyłączane do kompleksu. Etap ten jest poprzedzony przez wstępną
reakcję, w której inicjatorowi tRNA oddziałuje z IF2 tworząc tzw.
Kompleks podwójny. IF2 stymuluje wiązanie się inicjatorowego tRNA z
miejscem P. Kompleks IF2- inicjacyjny tRNA oraz mRNA zostają
następnie przyłączone do małej podjednostki w taki sposób, że kodon
inicjujący AUG cząsteczki mRNA jest umiejscowiony w środku miejsca
P (miejsca peptydowego) na małej podjednostce.

Pierwszym aminokwasem łańcucha polipeptydowego zarówno u
prokariotów jak i u eukariotów jest metionina. Jednak u prokariotów
metionina niesiona przez inicjatorowy tRNA jest zmieniana do
formylometioniny, przez dodanie grupy formylowej, która blokuje
grupę aminową aminokwasu
Końcową reakcją w inicjacji jest przyłączenie dużej podjednostki (50S)
Po przyłączeniu dużej podjednostki wszystkie czynniki inicjacyjne
odłączają się od rybosomu. Uwolnieniu IF2 towarzyszy hydroliza GTP.
Utworzony kompleks inicjacyjny zawiera teraz:
-rybosom
-mRNA
-fMet-tRNA, z inicjatorowym tRNA przyłączonym w miejscu P
rybosomu, naprzeciwko kodonu inicjującego AUG

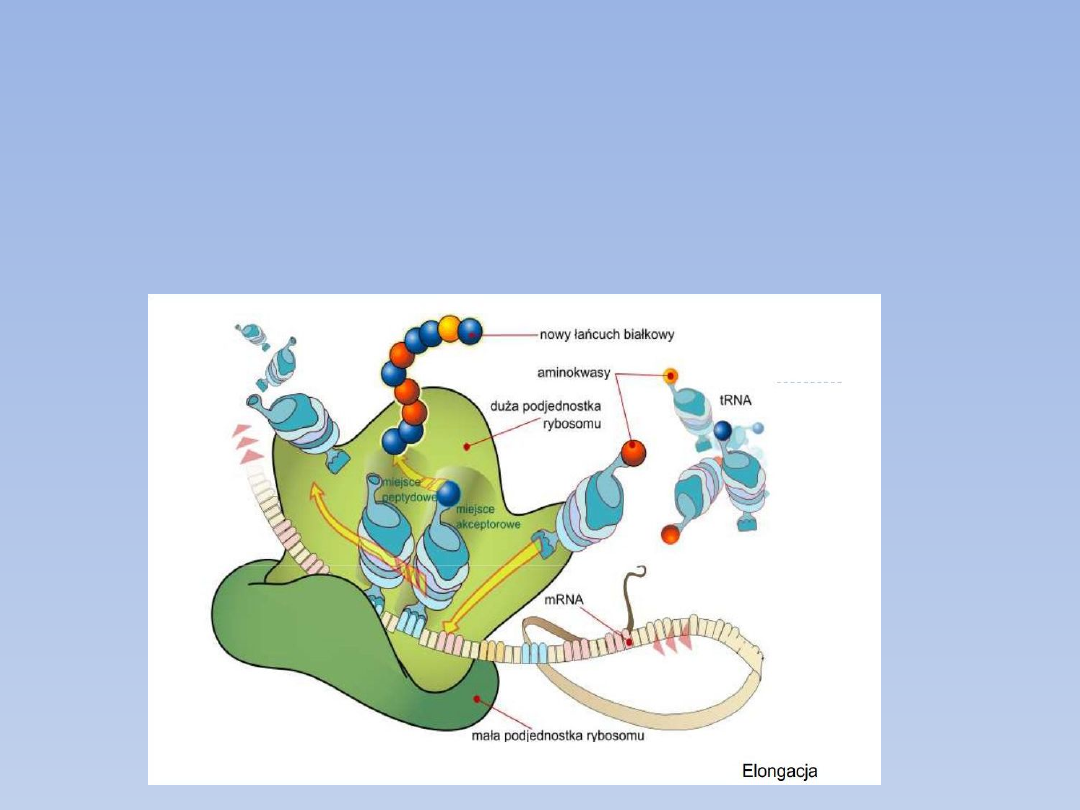
Elongacja

Elongacja ma miejsce, kiedy następny aminoacylo-tRNA przyłącza się
do rybosomu w miejscu A
Proces elongacji dzieli się na 3 główne etapy:
-wiązania aminoacylo-tRNA
-tworzenia wiązania peptydowego
-translokację.
Wszystkie te etapy są powtarzalne dla każdego aminokwasu
wyznaczanego przez kod genetyczny zawarty w mRNA aż do
wystąpienia sygnału zakończenia. Główne cechy tego procesu są takie
same jak u prokariotów i eukariotów, natomiast istnieją różnice w
szczegółach.
Wiązanie aminoacylo-tRNA:
Po utworzeniu kompleksu inicjacyjnego, formylometionylo-tRNA
(metionylo-tRNA u eukariotów) zajmuje w rybosomie wyjątkowo miejsce
peptydowe, miejsce zaś aminoacylowe jest jeszcze wolne Oba obszary
wiązania tRNA są umiejscowione na większej podjednostce (50S)
rybosom 70S (nadal kontynuowany będzie opis szeroko badanego
systemu prokariotycznego) Miejsce P na rybosomie zajęte jest przez
fMet-tRNA w 70S kompleksie inicjacyjnym. Drugi aminoacylo-tRNA
przyłącza się do miejsca A. Trójka zasad tRNA tworzy wiązania
wodorowe z trypletem zasad mRNA. Poza tym do związania cząsteczek
aminoacylo-tRNA w miejscu A niezbędny jest GTP oraz dwa białkowe
czynniki elongacyjne, EF-Tu i EF-Ts.

Czynnik elongacyjny Tu związany z GTP umieszcza aminoacylo-tRNA w
miejscu A, GTP ulega hydrolizie i kompleks EF-Ts łączy się z czynnikiem
EF-Tu, indukując równocześnie dysocjację GDP z kompleksu. Dalej GTP
wiąże się z czynnikiem EF-Tu, uwalniając przyłączony EF-Ts. Dopiero
teraz EF-Tu związany z GTP jest gotowy do przyłączenia następnego
aminoacylo-tRNA i dostarczenia go do miejsca A rybosomu. Szybkie
związanie aktywnego zaktywowanego tRNA przez czynniki EF-Tu ochroni
to wiązanie przed hydrolizą.
Gdy aminoacylo-tRNA znajduje się w miejscu A, a fMet-tRNA w miejscu P
rybosomu oraz gdy czynniki EF-Tu opuści rybosom, w kompleksie
znajduje się wszystko co jest potrzebne do utworzenia wiązania
peptydowego.
Wiązanie peptydowe jest tworzone w reakcji katalizowanej przez enzym
transferazę peptydową, która jest integralnym składnikiem podjednostki
50S
Reakcja tworzenia wiązania peptydowego:
Tworzenie wiązania zachodzi w centrum katalitycznym transferazy
peptydowej. Wydłużający się łańcuch peptydowy jest przenoszony na
grupę aminową wprowadzonego aminoacylo-tRNA. Po utworzeniu
wiązania peptydowego w miejscu A znajduje się dipeptydylo-tRNA,
natomiast w miejscu P- „pusty” tRNA, bez przyłączonego aminokwasu
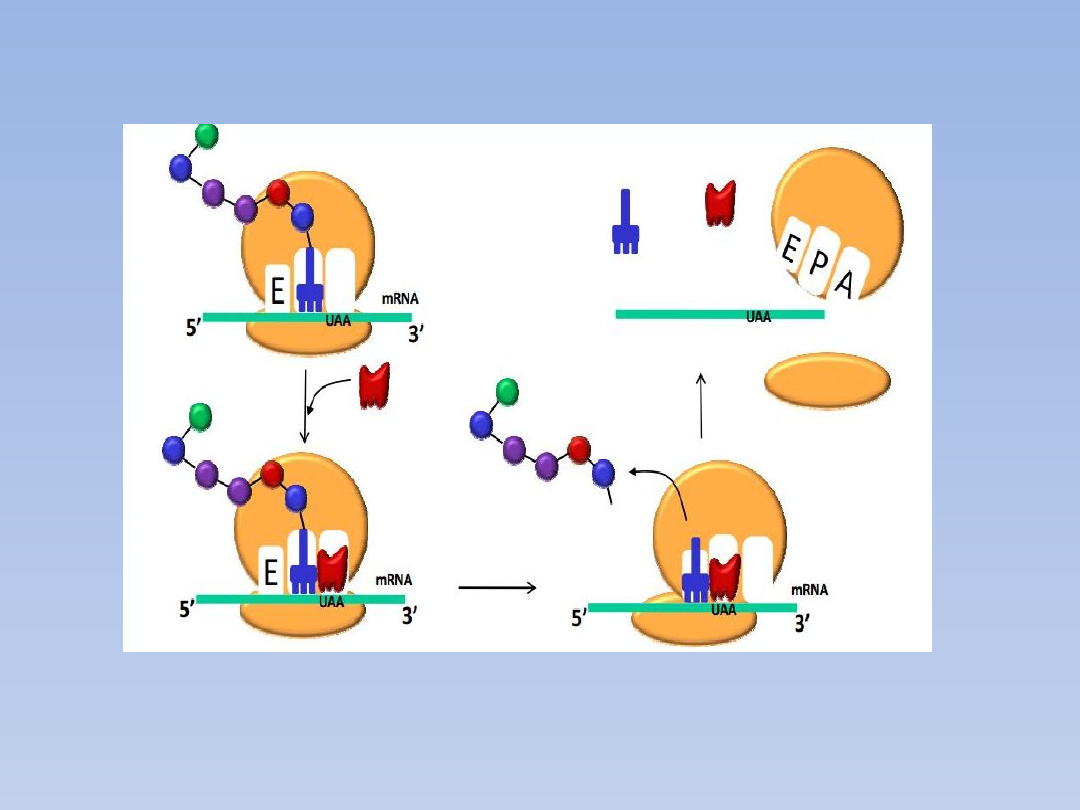
Translokacja:
W tym procesie nianaładowany tRNA jest uwalniany z miejsca P, a mRNA
przesuwa się o długość trzech nukleotydów.
Translokacja wymaga trzeciego czynnika elongacyjnego EF-G, który jest
również białkiem, i jeszcze raz GTP jest hydrolizowane do GDP i P
i .
Po translokacji miejsce A jest puste, gotowe do wiązania nowego
aminoacylo-tRNA i rozpoczęcia kolejnego cyklu elongacyjnego.

Terminacja

W terminacji łańcuch polipeptydowy zostaje uwolniony do
cytoplazmy.
Sygnał terminacji zakańcza biosyntezę białek. Takimi sygnałami są
kodony terminacyjne (nonsensowne) UAA, UAG i UGA (inaczej też
kodony STOP)
Z kodonem wiąże się bezpośrednio białkowy czynnik uwalniający
(RF-1 lub RF-2) oraz GTP, który jest związany z trzecim czynnikiem
uwalniający m, RF-3. RF-1 wiąże się z UAA i UAG, podczas gdy RF-2
wiąże się z UAA i UGA. RF-3 nie łączy się z żadnym kodonem, lecz
ułatwia aktywność pozostałych dwóch czynników uwalniających.
Zarówno RF-1, jaki i RF-2 (ang. Release factor; skrót – RF) wiążą się
w okolicach miejsca A rybosomu, gdy pojawi się w nim jeden z
kodonów terminacyjnych. Czynnik uwalniający nie tylko blokuje
wiązanie się nowego aminoacylo-tRNA ale również wpływa na
aktywność transferazy peptydowej, co powoduje hydrolizę wiązania
pomiędzy peptydem a tRNA zajmującym miejsce P. W wyniku tej
hydrolizy uwalnia się z miejsca P łańcuch polipeptydowy i tRNA. Po
odhydrolizowaniu łańcucha peptydowego od tRNA również rybosom
dysocjuje na podjednostki, które są dalej wykorzystywane w
następnych cyklach syntezy białka.
U Eukariota wszystkie funkcje wypełnia tylko jeden dimeryczny
czynnik eRF.
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
Wyszukiwarka
Podobne podstrony:
33 Przebieg i regulacja procesu translacji
33 Przebieg i regulacja procesu translacji
32 Proces i regulacja procesow translacji
BIOCHEMIA Przebieg procesu translacji
3 Charakterystyka i przebieg procesu translacji
BIOCHEMIA Przebieg procesu translacji
1 hormonalna regulacja procesów życiowych
Hormonalna regulacja procesów rozwojowych, BIOMEDYKA
Regulacja Procesów Fizjologicznych
Organizacja i przebieg przykładowego procesu produkcji, różne
Regulacja procesĂłw syntezy i wydzielania hormonĂłw tarczycy, Patomorfologia
kontrola i regulacja procesów życiowych GRUPA2 wer 2007, sprawdziany, gim1
kontrola i regulacja procesów życiowych, sprawdziany, gim1
Przebieg projektowania procesu technologicznego
kontrola i regulacja procesów życiowych GRUPA2, sprawdziany, gim1
37 REGULACJA PROCESOW ZYCIOWYCH RO
2 Biosynteza FA przebieg, regulacja
kontrola i regulacja procesów życiowych wer. 2007, sprawdziany, gim1
więcej podobnych podstron