1. 1 i 2 prawo Mendla, czego nie opisał Mendel w 2 prawie?
1 prawo Mendla: „Każda gameta zawiera tylko jeden gen z danej pary alleli.” (prawo czystości gamet)
2 prawo Mendla: „Geny należące do jednej pary alleli dziedziczą się niezależnie od genów należących
do drugiej pary alleli.” (prawo niezależnego dziedziczenia cech) Mendel nie wziął pod uwagę genów
sprzężonych znajdujących się na tym samym (homologicznym) chromosomie – opisał to zjawisko
dopiero T. Morgan.
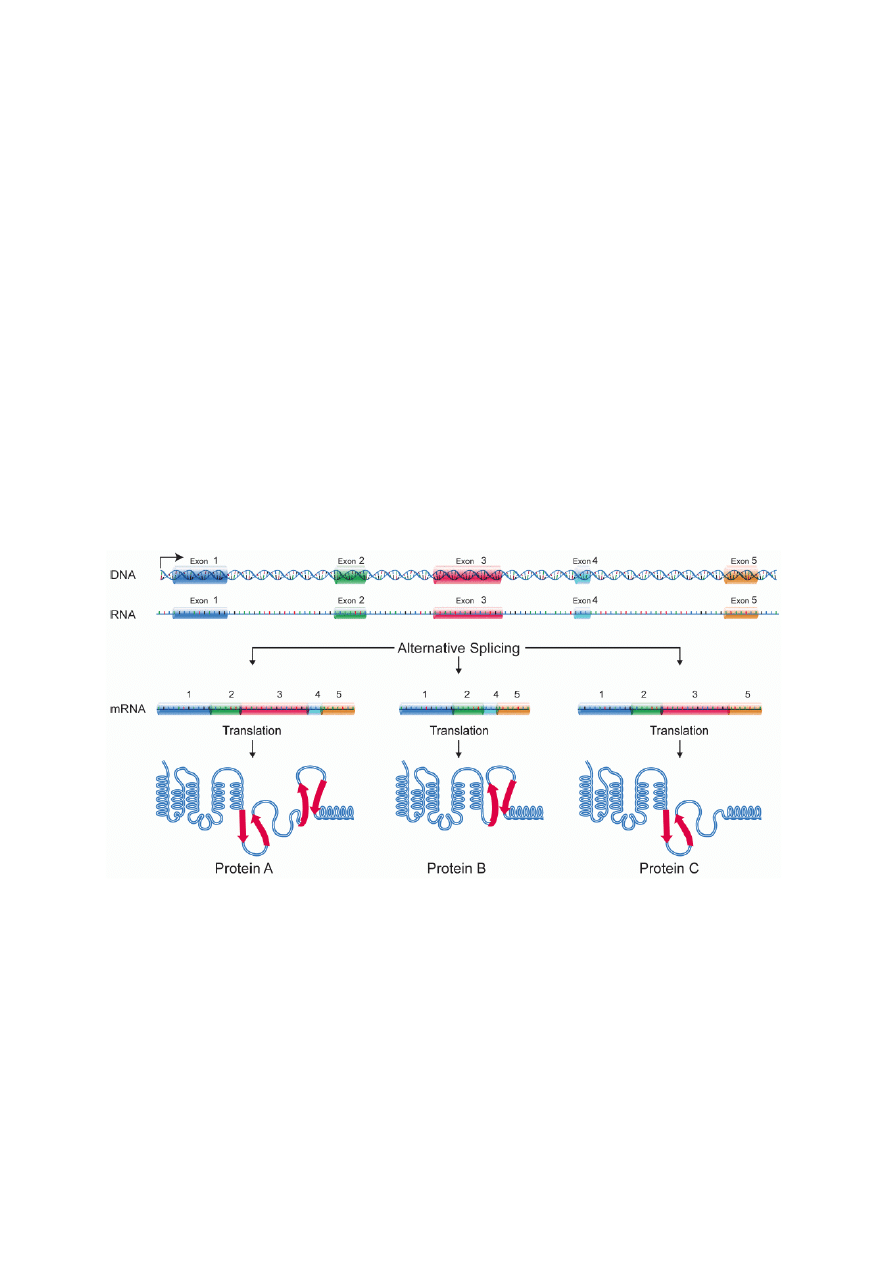
2. Alternatywny splicing.
Alternatywny splicing (alteratywne składanie egzonów) prowadzi do powstania różnych izoform
białka.
Istnieją cztery drogi alternatywnego splicingu:
•
wykorzystanie różnych promotorów podczas transkrypcji genu.
•
wykorzystanie różnych sygnałów do poliadenylacji
•
zachowywanie niektórych intronów
•
pomijanie niektórych egzonów.
3. Opisz terminacje translacji.
Syntezę łańcucha peptydowego kończą tzw. czynniki uwalniające, które rozpoznają kodony
terminacyjne (kodony „stop” – UAA, UGA, UAG) niekodujące żadnego aminokwasu. Sygnałem do
zakończenia translacji jest sytuacja, gdy jeden z kodonów STOP znajdzie się w miejscu A rybosomu.
Kodony STOP nie są rozpoznawane przez żadne tRNA, tylko przez czynniki uwalniające, które są
odpowiedzialne za hydrolizę wiązania estrowego peptydylo—tRNA i uwolnienie nowo powstałego
białka z rybosomu. U Prokaryota za proces terminacji translacji odpowiedzialne są dwa czynniki —
RF1 i RF2. Czynnik RF1 rozpoznaje kodony UAA i UAG, a RF2 rozpoznaje kodony UAA i UGA. U
Eukaryota jest tylko jeden czynnik uwalniający, który rozpoznaje wszystkie kodony terminujące
translację.
4. Podjednostka alfa i beta polimerazy III?
Kompleks polimerazy III zbudowany jest z podjednostek:
alfa – synteza DNA, aktywność katalityczna
beta – spinają enzym na nici DNA, tworzy klamrę na obu niciach, odpowiada za procesywność
enzymu
gamma – wprowadza beta na miejsce działania
tau – tworzenie dimeru na nici wiodącej, odpowiada za procesywność enzymu
epsilon – 3'-5'-nukleaza korekcyjna
teta – kompletowanie holoenzymu
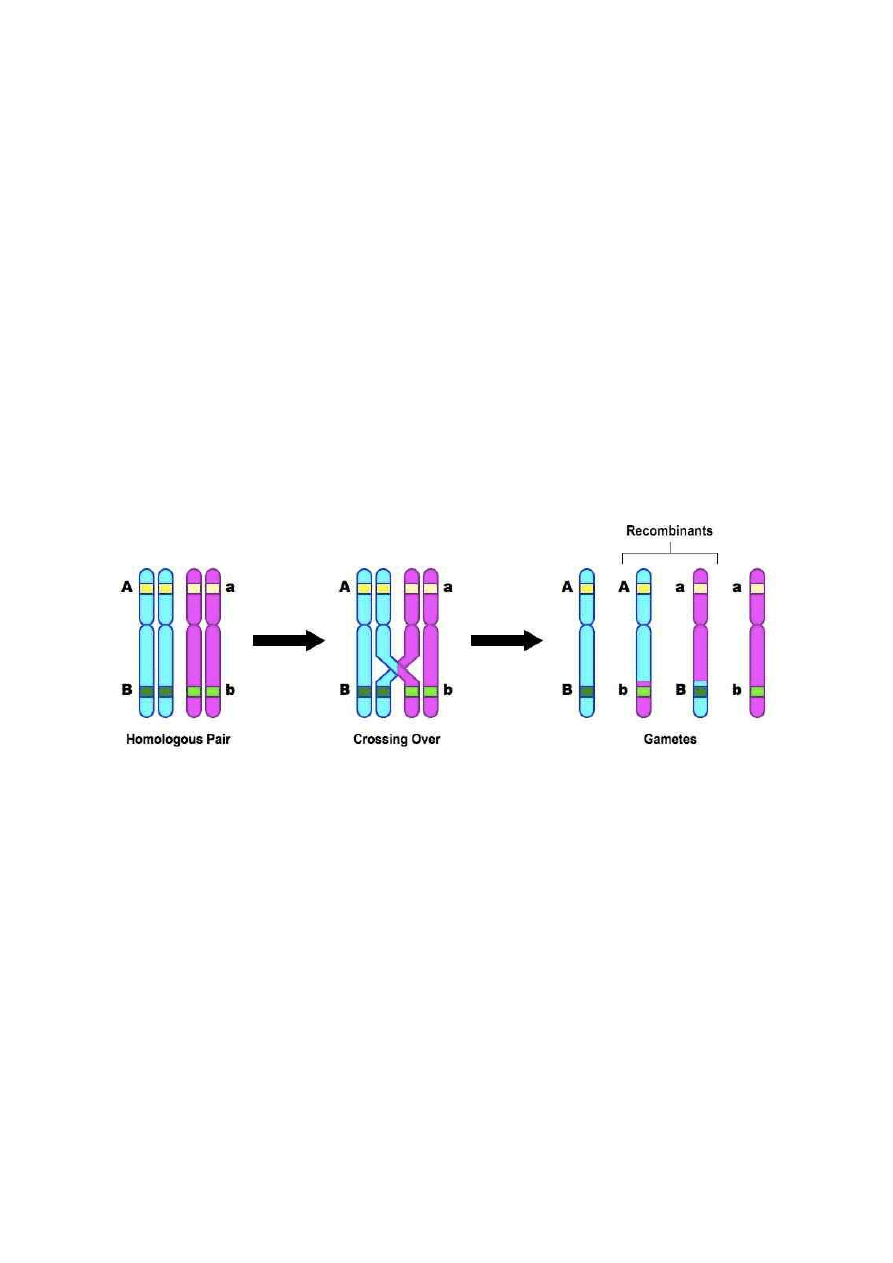
5. Jakie zjawisko opisał Morgan?
Morgan opisał zjawisko crossing-over.
6.Czy organizmy prokariotyczne rozmnażają się przez mitozę?
Komórki te NIE mogą się dzielić mitotycznie ani mejotycznie. Rozmnażają się przez prosty podział
komórki (amitoza). Do komórek prokariotycznych należą komórki bakterii (w tym sinic) oraz
archeonów.
Do wersji 3.
Bylo jeszcze jaka sekwencja terminuje transkrypcje.
Sekwencja terminatorowa (terminator) – sekwencja rozpoznawana przez polimerazę jako koniec
transkrypcji (odwrotne powtórzenia na końcu ORFu, tworzące strukturę hairpin).
Jak działa terminator transkrypcji? I jeszcze, jaką część stanowią u prokariota części kodujące białka.
Terminator - sekwencja kończąca, wyznaczającej miejsce terminacji (zakończenia) transkrypcji.
Sekwencja taka tworzy strukturę szpilki do włosów (hairpin), która zatrzymuje polimerazę RNA, co
powoduje rozpad kompleksu enzym - DNA - RNA. Drugim mechanizmem terminacji

wykorzystywanym przez bakterie jest terminacja Rho-zależna, gdzie do terminacji transkrypcji
potrzebne jest działanie czynnika Rho (ρ). Jest to białko zbudowane z 6 podjednostek (3*2), tworzące
pierścień naokoło mRNA (aktywny jako ATPaza i helikaza – hydrolizuje wiązania wodorowe). Białko
wiąże się w tzw. Rho-site i porusza się za polimerazą, a dogania jąw miejscu terminatora (ponieważ
RNAP zwalnia na sekwencjach palindromowych). Tam aktywności helikazowa białka Rho uwalnia
transkrypt i następuje terminacja. Transkrypt prokariotyczny nie wymaga dalszej obróbki. U bakterii
translacja rozpoczyna się, zanim transkrypcja dobiegnie końca. U Eukaryota procesy te rozdzielone są
w czasie i przestrzeni.
U prokariotów niemal cały operon stanowi materiał genetyczny kodujący białka. W skład
pojedynczego operonu wchodzą:
•
geny kodujące białka i enzymy (geny struktury lub też geny strukturalne),
•
dwa odcinki DNA niekodujące białek: promotor i operator.
•
niekiedy geny regulatorowe (np. enhancery)
Wersja 4:
1. Co to jest polimorfizm
Polimorfizm - w genetyce oznacza występowanie różnic w DNA populacji. Polimorfizmem nie określa
się jednak zmian rzadkich. Kryterium zaliczenia do tej kategorii jest, aby dana zmiana była częstsza niż
1% (innymi słowy zbyt częsta, by można było mówić o mutacji).
Polimorfizm można podzielić na:
- SNP (ang. Single Nucleotide Polymorphism) - polimorfizm pojedynczego nukleotydu;
- polimorfizm sekwencji mini - i mikrosatelitarnych.
Polimorfizm może odnosić się do sekwencji kodujących lub niekodujących. Polimorfizm genowy
oznacza występowanie różnorodnych odmian danego genu, co w konsekwencji może prowadzić do
różnic w budowie i działaniu białka kodowanego przez ten gen. Przykładem jest podatność na
działanie alkoholu etylowego wśród rasy azjatyckiej, związana z różnicą w budowie enzymu
dehydrogenazy alkoholowej. Natomiast zmiany w sekwencjach niekodujących mogą prowadzić do
zmiany ekspresji białek.
Zjawisko polimorfizmu pojedynczych nukleotydów wykorzystuje się w technice RFLP (ang. Restriction
Fragments Lenght Polymorphism).
2. Za co odpowiada LexA
Białko LexA jest represorem wszystkich operonów SOS, ystępuje jako homodimer (zbudowany z
domeny dimeryzacyjnej i domeny wiążącej DNA). Gdy system naprawy SOS jest wyłączony następuje
ekspresja genu lexA, dzięki czemu powstaje białko LexA. Białko to wiąże się z sekencjami promotorów
SOS, blokując ich ekspresję (dezaktywuje system SOS) i z promotorem genu recA, w ten sposób
modulując stężenie białka RecA w komórce. Włączenie systemu SOS stanowi sygnał dla białka RecA

(obecnego w komórce przez cały czas w niewielkich ilościach) do nieodwracalnego przecięcia dimeru
LexA, co umożliwia ekspresję genów systemu SOS.
3. Za co odpowiada DnaA
DnaA to inicjatorowe białko, które wiąże się do dsDNA (double-stranded DNA = dwuniciowego) w
miejscu ori i używając energii ATP, w obecności białek DnaB i DnaC rozwija nieznacznie DNA, tworząc
oczko replikacyjne.
5. Czemu telomery są ważne dla organizmów
Telomery to „czapeczki” bogate w pary GC, tworzone są dla ochrony końców chromosomów, złożone
z wielu tandemowych powtórzeń specyficznych gatunkowo sekwencji. Ludzkie telomery mają
sekwencję bogatą w G (GGGTTA) i rozciągają się na długości ok. 10 000 nukleotydów.
7. Jaka jest aktywność transpozazy
Transpozaza – enzym (endodeoksyrybonukleaza) rozpoznający sekwencje DNA na końcu transpozonu.
Transpozaza wiąże się z DNA i przecina go, uwalniając transpozon, a następnie przecina docelową
cząsteczkę DNA i przyłącza do niej transpozon.
Jeszcze do wersji 3: jak zbudowany jest transpozon Tn5.
Transpozon zbudowany z 5700 pz, występujący u E. coli w plazmidzie R, koduje gen oporności na
kanamycynę (i streptomycynę).
DO 4: O czym mówią prawa Chargaffa.
Reguła Chargaffa - prawidłowość dotycząca składu ilościowego zasad azotowych nukleotydów w
dwuniciowym DNA odkryta przez Erwina Chargaffa w latach 1950-1952. Głosi ona, że stosunki
adeniny (A) do tyminy (T) oraz cytozyny do guaniny (G) wynoszą zawsze ok. 1:1. Regułę tę można
opisać następującymi równaniami:
%A/%T = %C/%G = 1
%A = %T i %C = %G

Dla %A/%G, %A/%C i %G/%T nie obserwuje się podobnej zależności.
Reguła Chargaffa była jedną z podstawowych przesłanek, które pozwoliły Watsonowi i Crickowi na
opracowanie poprawnego modelu struktury DNA. Przyczyną prawidłowości zaobserwowanych przez
Chargaffa jest zasada komplementarności.
Zasada komplementarności (komplementarność - łac. complementum - dopełnienie, uzupełnienie) –
zasada wzajemnego uzupełniania się, np. topologie komplementarne, dobra komplementarne,
komplementarne gwinty, antonimy komplementarne. W stereochemii pojęcie jest stosowane
najczęściej w odniesieniu do struktur DNA, powstających dzięki komplementarności struktur par
zasad, występujących w sąsiednich niciach DNA lub DNA i RNA.
Zgodnie z zasadą komplementarności cytozyna (C) łączy się tylko z guaniną (G).
Adenina (A) w kwasie RNA łączy się z uracylem (U), a w kwasie DNA łączy się z tyminą (T). Na
podstawie tej zasady możliwe jest odtworzenie brakującej nici DNA, np. podczas replikacji.
Zasada komplementarności dla transkrypcji RNA na DNA przedstawia się w następujący sposób :
•
Adenina (A) ←→ Uracyl (U); - w RNA
•
Tymina (T) ←→ Adenina (A);
•
Guanina (G) ←→ Cytozyna (C);
-----------------------------------
wersja 2:
2. Czy wysoka temp uszkadza dna
Szoki temperaturowe mogą powodować hydroizę wiązania beta-N-glikozydowego łączącego zasadę z
cukrem i powstanie miejsca AP (apurynowego lub apirymidynowego), czyli pozbawionego zasady. Na
dwuniciowej cząsteczce DNA powstaje luka, która zwykle jest naprawiana i nie powoduje mutacji (u
ludzi dziennie powstaje ok. 10 000 miejsc AP). Do mutacji może dojść wówczas, gdy w komórce
włączony jest system naprawy SOS, który wszystkie luki wypełnia adeniną, bez względu na nukleotyd
nici komplementarnej.
3. Znaczenie enzymow restrykcyjnych:
Są trzy systemy restrykcji, o których jest napisane w artykule, który podrzuciłem w komentarzu.
4. Jak rozumiesz pojecie chromosomy homologiczne i chromosomy niehomologiczne?
Wydaje mi się, że chodzi o ich podobieństwo. Chromosomy homologiczne mają w tych samych
miejscach locus tych samych genów (mogą być różne allele). Oprócz tego łączy je także podobieństwo
wyglądu (nie ma możliwości, by chromosom metacentryczny i akrocentryczny były homologicznymi,
ponieważ mieszczą różną ilość informacji genetycznej).
5. Jaka cześć genomu ludzkiego koduje białka?
Projekt poznania ludzkiego genomu (eng. Human Genome Project) był to program naukowy mający
na celu poznanie sekwencji wszystkich par komplementarnych tworzących ludzki genom, zawierający
ok. 20-25 tys. genów. Według twórców bazy danych PANTHER ludzki genom ma ok. 18 tys genów
kodujących białka. Część kandydatów okazała się pseudogenami, część fragmentami innych genów.

Poza tym nasz genom zawiera liczne geny kodujące RNA (rRNA, miRNA, tRNA, piRNA), które nie
kodują białek, sekwencje regulatorowe, sekwencje repetytywne, centromerowe, telomerowe,
elementy ruchome, a także nieaktywne już pseudogeny. Duże fragmenty genomu można usuwać bez
szkody dla myszy, więc należy założyć, że u ludzi jest podobnie, i moglibyśmy funkcjonować, gdyby
nasze genomy miały znacznie mniej, niż te 3,1 mld par zasad.
6. Enzymy w transpozycji:
Fotoreaktywacja:
Fotoreaktywacja - jeden z mechanizmów naprawy DNA, w którym enzym fotoliaza specyficznie
reaguje z jedno- lub dwuniciowym fragmentem DNA, zawierającym fotodimery pirymidyn (tyminowe,
cytozynowe i cytozynowo-tyminowe). Pod wpływem światła o długości fali 310-450 nm, następuje
enzymatyczne rozszczepienie fotodimer|fotodimerów do monomerów i przywrócenie DNA do stanu
pierwotnego. Mechanizm naprawy przez fotoreaktywację jest bezbłędny, nie prowadzi do mutacji.
Proces fotereaktywacji zachodzi bezustannie w organizmie, w komórkach narażonych na działanie
promieni UV. Enzym fotoliaza odpowiedzialny za fotoreaktywację został wykryty u bakterii (m.in.
Escherichia coli), grzybów, roślin i niektórych kręgowców (brak enzymu u człowieka i innych
łożyskowców).
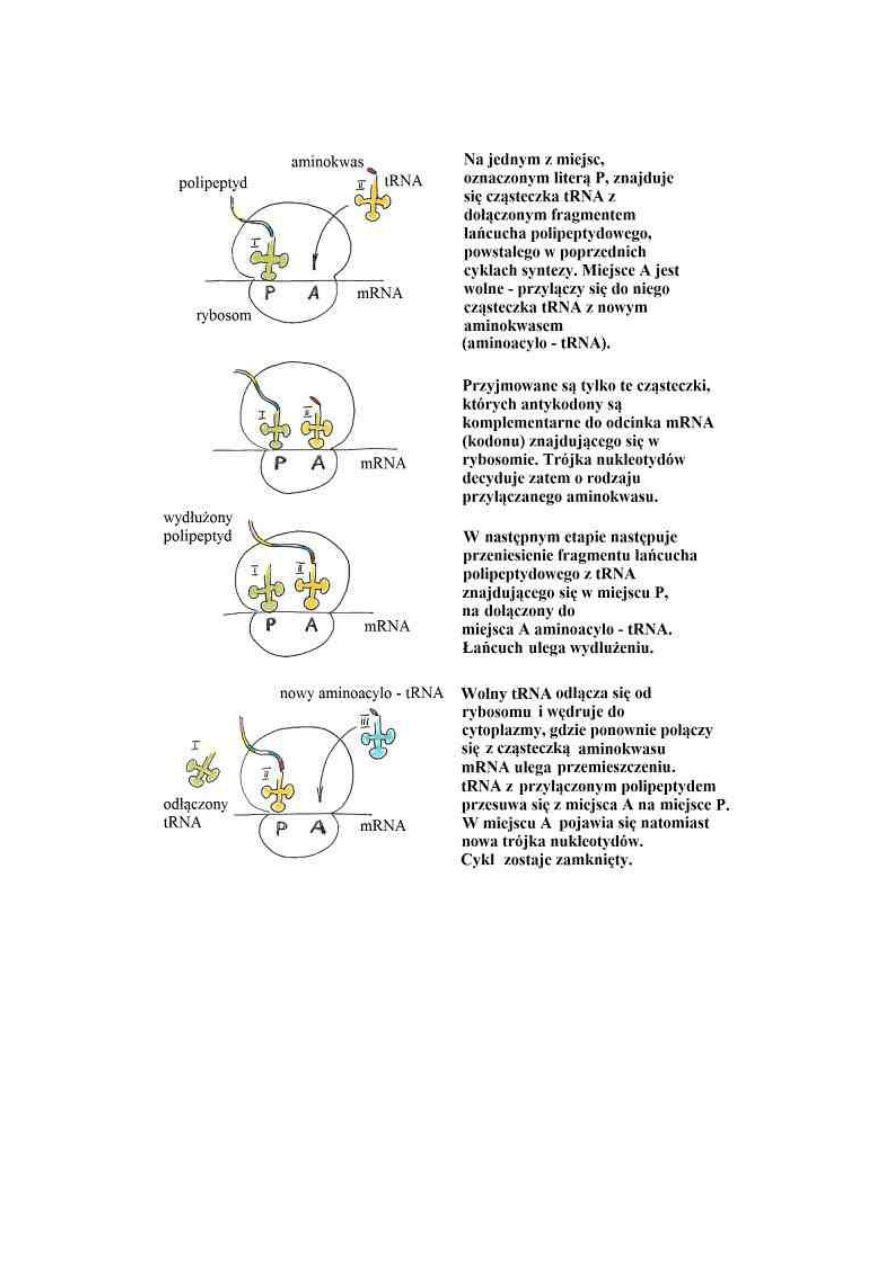
Elongacja translacji:
Dlaczego orf latwiej odczytujemy u prok niż u eukariota?
Otwarta ramka odczytu lub ORF (z ang. Open Reading Frame) jest każdą sekwencją DNA lub RNA,
która potencjalnie może ulec translacji na białko. W genie ORF mieści się pomiędzy kodonem START, a
kodonem STOP. ORF-y odnajduje się zwykle podczas przeczesywania sekwencji DNA przy
poszukiwaniu potencjalnych genów. Dla różnych organizmów, z powodu różnorodności sekwencji
START istnieją zatem różne ORF-y. Typowy algorytm poszukiwania ORF uwzględni zatem kod
genetyczny danego organizmu (łącznie z możliwymi jego odmianami) i wyszuka wszelkie możliwe ORF
(z czego nie wszystkie muszą być prawidłowe).

W rzeczywistości, obecność ORF, szczególnie długiego, jest zazwyczaj dowodem na obecność genu,
gdzieś w okolicznej sekwencji. W takim wypadku ORF jest częścią sekwencji kodującej, podlegającej
translacji przez rybosomy. Jednak można spotkać także ORF-y nie będące częścią genu - zazwyczaj są
one krótkie i kończą się zaledwie po kilku kodonach.
Po sekwencjonowaniu genu jest bardzo ważne, żeby wyznaczyć jego ORF. Dla organizmów z
dwuniciowym DNA jego sekwencja może być odczytana na sześć różnych sposobów - trzy na nici
wiodącej i trzy na nici opóźnionej. Zazwyczaj najdłuższa sekwencja, nie przerwana kodonem STOP w
obrębie genu jest jego ORF-em. Jakkolwiek sprawdza się to dla prokariota, u eukariota sprawa
komplikuje się, gdyż ich geny zawierają długie sekwencje niekodujące - introny. ORF dla takich genów
odnajduje się badając zazwyczaj mRNA będące ich produktem.
Aneuploid przyczyny:
Aneuploidia jest jednym z kilku zaburzeń ilości materiału genetycznego i oznacza, że jego ilość w
komórkach nie jest wielokrotnością zawartości DNA w jednym garniturze chromosomów typowym dla
danego osobnika lub gatunku (euploidia), ale ma wartość pośrednią.
Przyczyną tej mutacji jest nondysjunkcja polegająca na niewłaściwym rozejściu się (braku rozejścia)
chromosomów w czasie mejozy. Konkretnie dzieje się to w procesie anafazy. Główną przyczyną
nondysjunkcji jest nieprawidłowo wykształcone wrzeciono podziałowe. Czynnikiem mutagennym
chemicznym, który prowadzi do zaburzeń w prawidłowym wytwarzaniu wrzeciona podziałowego jest
przede wszystkim kolchicyna.
Triploid - u człowieka, poliploidia polegająca na obecności w komórce dodatkowego zestawu
chromosomów (zapis kariotypu 69,XXY, 69,XXX albo 69,XYY). Triploidia może dotyczyć wszystkich
komórek organizmu albo tylko w części (mozaicyzm).
Monoploid - organizm, w którego komórkach somatycznych występuje jeden zespół chromosomów
niehomologicznych, nazywany podstawowym zespołem chromosomów. Liczbę chromosomów
zespołu podstawowego określa się liczbą podstawową (23, X). U monoploidów jest ona równa liczbie
haploidalnej.
Przyczyna hemofilii i albinizmu:
Hemofilia A i B są chorobami sprzężonymi z płcią. Geny, których mutacje wywołują choroby, znajdują
się na chromosomie X. Hemofilie dziedziczone są w sposób recesywny, co oznacza, iż chorują jedynie
osoby z pełną ekspresją recesywnego genu.
Albinizm wywołany jest przez brak enzymu tyrozynazy przekształcającego prekursor melaniny w
barwnik melaninę. Warunkuje go gen recesywny. Oznacza to, że jeśli oboje rodzice są
heterozygotyczni pod względem genu aktywności tyrozynazy to ich dziecko z 25%
prawdopodobieństwem będzie albinosem.
Budowa BER lub NER
Polecam wrzuconą prezentację i książkę Sadakierskiej (od str. 107).
Różnica miedzy mapą fizyczną a genetyczną
Mapa genetyczna - mapa lokalizacji genów na chromosomie, powstała poprzez badanie częstości
zachodzenia zjawiska crossing over (rekombinacji) pomiędzy odpowiednimi genami na podstawie
stosunku ilości rekombinantów do ilości osobników nie zrekombinowanych otrzymanych w krzyżówce
testowej. Im częściej zachodzi crossing over między genami (im większa jest wartość tego stosunku),
tym bardziej są one od siebie oddalone. Odległość tą wyraża się w jednostkach mapowych (j.m.) lub
w centymorganach (cM).
Mapa genetyczna - schemat uporządkowania miejsc genowych w obrębie chromosomów. Mapy
fizyczne różnią się od map genetycznych tym, że ich konstrukcja jest oparta na długości fragmentów
restrykcyjnych i sekwencjonowaniu DNA; skala map fizycznych to liczba par zasad dzielących geny.
Mapy genetyczne porządkują geny w sposób orientacyjny, podając odległości w oparciu o przybliżoną
miarę frakcji rekombinacji.
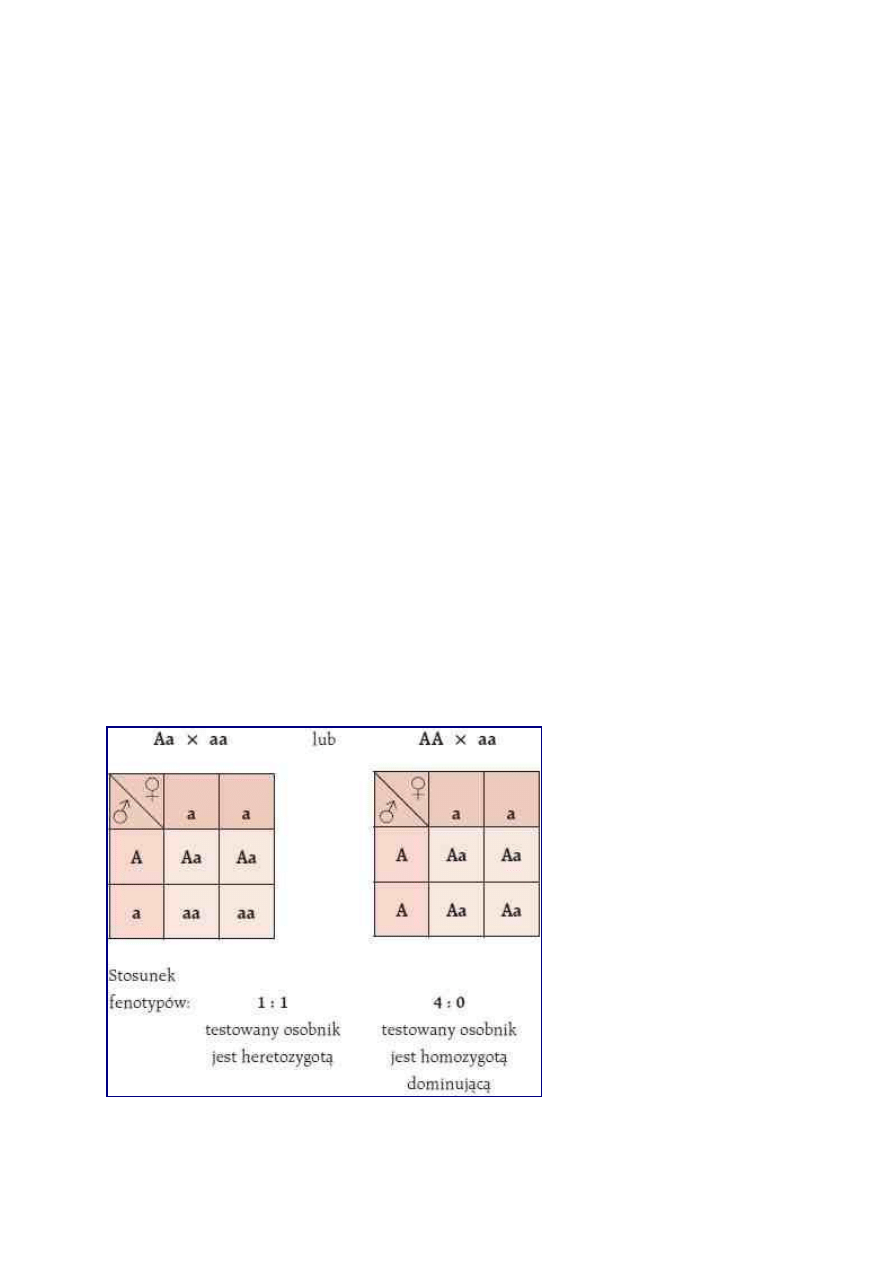
Dlaczego krzyżówkę testową wykonuje się z homozygota recesywna?
Ponieważ krzyżówka testowa ma nam odróżnić hetoerozygotę od homozygoty dominującej w
przypadku pełnej dominacji.

Jaka jest ilość w przybliżeniu transpozonów u człowieka (chyba jakoś tak)?
Na oko, chyba z pół metra. Nie znalazłem żadnych danych (ani procentowych, ani liczbowych). Jakby
ktoś miał pomysł, odsyłam do komentarzy.
Czy nawozy azotowe powodują mutacje?
Tak... Podobny problem jak wyżej. Ktoś ma uzasadnienie (nie wiem? Hydroksylamina? Dodatkowa
aminacja? Zwiększenie częstości zachodzenia tautomerii zasad azotowych? Kwaśne deszcze?), pisać w
komentarzach.
Czy geny kodujące rodzinę białek globulin pełnia te sama funkcje?
Czyż dwa różne geny nie pełnią dwóch różnych funkcji? Kolejne pytanie do dyskusji. Nie mam
pomysłu, z której strony je ugryźć? Może jak Suarez?
Wyszukiwarka
Podobne podstrony:
odpowiedzi na pytania 2 id 3325 Nieznany
ODPOWIEDZI NA PYTANIA 4 id 3325 Nieznany
Odpowiedzi na pytania id 332587 Nieznany
odpowiedzi do testu id 332437 Nieznany
projekt 04 01 10r na rm id 3979 Nieznany
podloga na gruncie id 364776 Nieznany
Na egzamin id 312078 Nieznany
Kompozyty na kolo id 243183 Nieznany
Odpowiedzi testu BHP id 332669 Nieznany
Igloo na zal id 69618 Nieznany
Operacje na macierzach id 33628 Nieznany
POZWOLENIA NA BRON id 380565 Nieznany
opracowanie na kolosa id 338294 Nieznany
Konspekt nr 5 na cw 6 id 245644 Nieznany
Na egzamin 2 id 312084 Nieznany
gotowa na sukces id 193702 Nieznany
gotowa na sukces 2 id 193703 Nieznany
najwazniejszy pdf na HES id 313 Nieznany
Konspekt nr 6 na cw 7 id 245645 Nieznany
więcej podobnych podstron