Podstawowe informacje o DNA i RNA
Struktura DNA umo
ż
liwia przechowywanie informacji oraz samopowielanie cz
ą
steczki
DNA został powszechnie uznany za materiał
genetyczny dopiero po opublikowaniu przez
Jamesa Watsona i Francisa Cricka pracy, w
której zaproponowali oni model budowy
cząsteczki DNA, wyjaśniający w błyskotliwy
sposób podstawowe problemy dotyczące
dziedziczności.
Okoliczności
związane
z
konstruowaniem tego modelu to jeden z
najwspanialszych
rozdziałów
historii
współczesnej biologii. Gdy Watson i Crick
zainteresowali się zagadnieniem struktury
DNA, wiele już wiedziano na temat jego
fizycznych
i
chemicznych
właściwości.
Szczególny
wkład
obu
uczonych
w
rozwiązanie
zagadki
DNA
polegał
na
powiązaniu ze sobą tych informacji i
niezwykle trafnym wykorzystaniu ich do
budowy modelu, który wyjaśniał, jakie cechy
umożliwiają cząsteczce DNA magazynowanie
informacji
genetycznej
i
jednoczesne
pełnienie
roli
matrycy
w
procesie
samoodtwarzania się DNA.
Nukleotydy ł
ą
cz
ą
si
ę
wi
ą
zaniami kowalencyjnymi w długie polimery, w których mog
ą
wyst
ę
powa
ć
w dowolnej kolejno
ś
ci
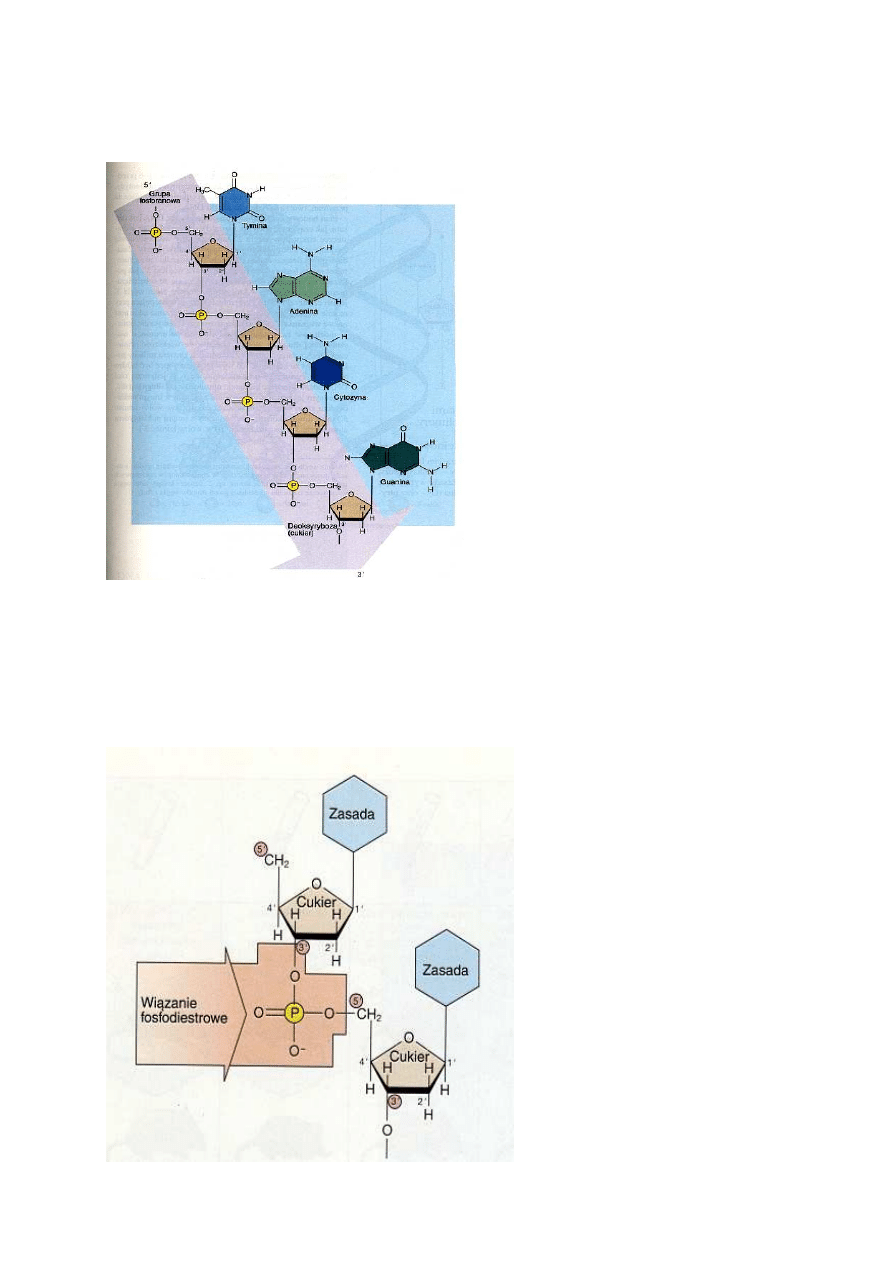
Jednostkami tworzącymi DNA są nukleotydy, składające się z cukru pentozy - deoksyrybozy, grupy
fosforanowej i zasady azotowej. Zasady występujące w DNA to: puryny - adenina (A) i guanina (G)
- oraz pirymidyny - tymina (T) i cytozyna (C). Połączone wiązaniami kowalencyjnymi nukleotydy
zawierają reszty fosforanowe i cząsteczki cukru, występujące na przemian, tworzące szkielet
cząsteczki DNA.
Plan
budowy
nukleotydowych
podjednostek DNA jest taki sam, jak
cząsteczki AMP - z tym tylko, że w
cząsteczce cukru przy węglu 2'
zamiast
grupy
wodorotlenowej
znajduje się atom wodoru (stąd
nazwa deoksyryboza). (Atomy węgla
w
cząsteczkach
numeruje
się
powszechnie
zgodnie
z
zasadą
stosowaną w chemii organicznej. W
chemii kwasów nukleinowych indeks
"prim" przy numerze atomu, np. 2',
oznacza kolejny atom węgla w
cząsteczce cukru dla odróżnienia go
od atomów węgla zasady.) Zasada
azotowa dołączona jest do węgla l'
cząsteczki cukru, natomiast reszta
fosforanowa połączona jest z węglem
5'. Nukleotydy łączą się ze sobą
wiązaniem kowalencyjnym, w którym
uczestniczą atom węgla 3' jednej
cząsteczki cukru i grupa fosforanowa
występująca przy atomie węgla 5'
drugiej cząsteczki cukru; wiązanie
takie nosi nazwę wiązania 3', 5'-fosfodiestrowego. Teoretycznie istnieje możliwość tworzenia
nukleotydowych polimerów o nieskończonej długości. Wiemy obecnie, że większość cząsteczek DNA
występujących w komórkach zawiera miliony nukleotydów i że kolejność ich występowania może
być bardzo różna. W pojedynczej nici DNA można wyróżnić kierunek; niezależnie jak długa jest nić,
na jednym jej końcu występuje nie związany z innym nukleotydem atom węgla 5' cząsteczki cukru
(tzw. wolny koniec 5'). a na drugim końcu - nie związany z innym nukleotydem atom węgla 3'
cząsteczki cukru (tzw. wolny koniec 3').
DNA zbudowany jest z dwóch oplecionych wokół siebie nici polinukleotydowych, które tworz
ą
podwójny heliks
Ważnych informacji o budowie DNA dostarczyły badania dyfrakcji promieni X (promienie rentgena)
na kryształach oczyszczonego DNA. Badania te prowadziła Rosalinda Franklin w laboratorium M. H.
F. Wilkinsa. Pomiar dyfrakcji promieni X stanowi bardzo skuteczną metodę wyznaczania odległości
między atomami w cząsteczkach ułożonych w regularne, powtarzające się struktury krystaliczne.
Długość fali promieni X jest tak mała, że są one rozpraszane przez elektrony, które otaczają
tworzące cząsteczkę atomy. Atomy o dużej gęstości elektronowej (np. fosfor, tlen) powodują
silniejsze ugięcie promieni X niż atomy zawierające mniejszą liczbę elektronów (tj. atomy o niższej
liczbie atomowej). Gdy kryształ zostaje naświetlony wiązką promieni X o dużej intensywności,
promienie te ulegają charakterystycznemu rozproszeniu (ugięciu) spowodowanemu regularnym
ułożeniem atomów w krysztale. Wzór rozpraszania promieni X wygląda na kliszy jak zbiór ciemnych
plamek. Analiza matematyczna ułożenia i odległości między plamkami umożliwia precyzyjne
określenie odległości między atomami i ich ułożenia w cząsteczce.
W okresie, gdy Watson i Crick
zaczęli zajmować się problemem
struktury
DNA,
Franklin
dysponowała już zdjęciami, które
ukazywały
klarownie
charakterystyczny dla DNA wzór
rozpraszania.
Zdjęcia
te
wskazywały jasno, że badana
struktura jest typu helikalnego
oraz
że
w
cząsteczce
DNA
występują
trzy
główne,
powtarzające
się
regularnie
elementy
struktury,
których
okresy powtarzalności wynoszą
odpowiednio 0,34 nm, 3,4 nm i
2,0
nm.
Franklin
i
Wilkins
wywnioskowali z tych danych, że
zasady (które są płaskie) ułożone
są w DNA jedna nad drugą w
postaci
stosu,
podobnie
jak
szczeble drabiny. Korzystając z tej
informacji
Watson
i
Crick
przystąpili
do
budowania
powiększonych
w
odpowiedniej
skali modeli składników DNA, a
następnie zaczęli dopasowywać je
do siebie tak, by uzyskać zgodność
z wynikami doświadczeń.
W wyniku szeregu prób dwaj
uczeni zbudowali wreszcie model
zgodny z istniejącymi danymi.
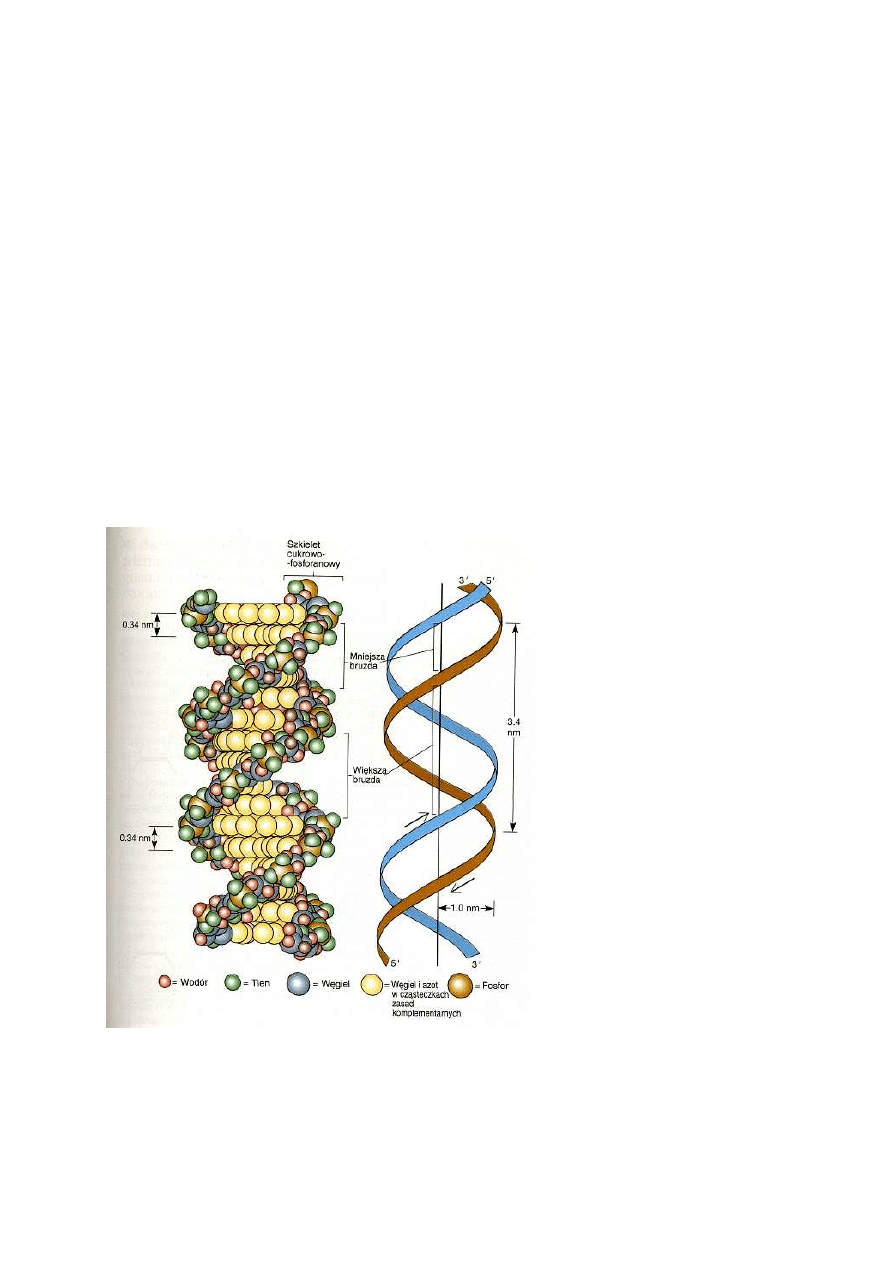
Wymiary cząsteczki odpowiadały tym, które wynikały z pomiarów dyfrakcji tylko wówczas, gdy DNA
zbudowany był z dwóch łańcuchów polinukleotydowych zwiniętych w podwójny heliks. W modelu
Watsona i Cricka szkielet fosforanowo-cukrowy dwóch łańcuchów przebiega po zewnętrznej stronie
heliksu. Zasady należące do dwóch leżących naprzeciw siebie łańcuchów tworzą pary wypełniające
środek struktury. Model ukazywał od razu przyczynę istnienia periodyczności 0,34 nm i 3,4 nm.
Każda para zasad oddalona jest dokładnie o 0,34 nm od pary leżącej nad nią i pary leżącej pod nią.
Ponieważ na jeden pełny skręt heliksu przypada 10 par zasad, długość każdego skrętu wynosi 3,4
nm. Aby zachować zgodność modelu z danymi doświadczalnymi, dwa łańcuchy tworzące cząsteczkę
DNA musiały przebiegać w przeciwnych kierunkach. Każdy koniec podwójnego heliksu musi mieć
zatem wolną resztę 5' fosforanową na jednym łańcuchu i wolną grupę 3' wodorotlenową na drugim.
Ponieważ łańcuchy polinukleotydowe w cząsteczce DNA przebiegają w przeciwnych kierunkach,
nazywa się je przeciwbieżnymi.
W dwuniciowym DNA pomi
ę
dzy adenin
ą
i tymina oraz pomi
ę
dzy guanin
ą
i cytozyn
ą
tworz
ą
si
ę
wi
ą
zania wodorowe
Inne założenia, które posłużyły do budowy modelu, uzyskano dzięki pomysłowemu powiązaniu
danych chemicznych z wynikami badań nad dyfrakcją promieni X. W 1950 r. Erwin Chargaff i jego
współpracownicy z Uniwersytetu Columbia oznaczyli skład zasad w cząsteczkach DNA
pochodzących komórek wielu różnych organizmów. Ustalili oni prostą prawidłowość o istotnym dla
budowy DNA znaczeniu. Niezależnie od źródła, z jakiego pochodził DNA, jak to sformuał Chargaff,
"ilościowy stosunek puryn do pirymidyn, a także adeniny do tyminy oraz guaniny do cytozyny jest
zawsze bliski jedności". Innymi słowy, w cząsteczce DNA A=T i G=C.
Na podstawie wyników badań dyfrakcji promieni X ustalono, że podwójny heliks ma ściśle
określoną i stałą szerokość, na co wskazywał okres powtarzalności 2,0 nm. Obserwacja ta ma
związek z regułą Chargaffa. Zauważmy, że pirymidyny (cytozyna i tymina) mają tylko po jednym
pierścieniu, są zatem mniejsze niż dwupierścieniowe puryny (adenina i guanina). Obserwacje
dokonane na modelach nasunęły Watsonowi i Crickowi myśl, że gdyby każdy "szczebel drabiny"
(łączący obie nici) składał się z jednej cząsteczki puryny i jednej cząsteczki pirymidyny, szerokość
heliksu wynosiłaby dokładnie 2,0 nm; w miejscu połączenia dwóch zasad purynowych (z których
każda ma szerokość 1,2 nm) odległość między nićmi musiałaby być większa niż 2,0 nm, a w
miejscach połączenia dwóch pirymidyn odległość musiałaby być mniejsza niż 2,0 nm.
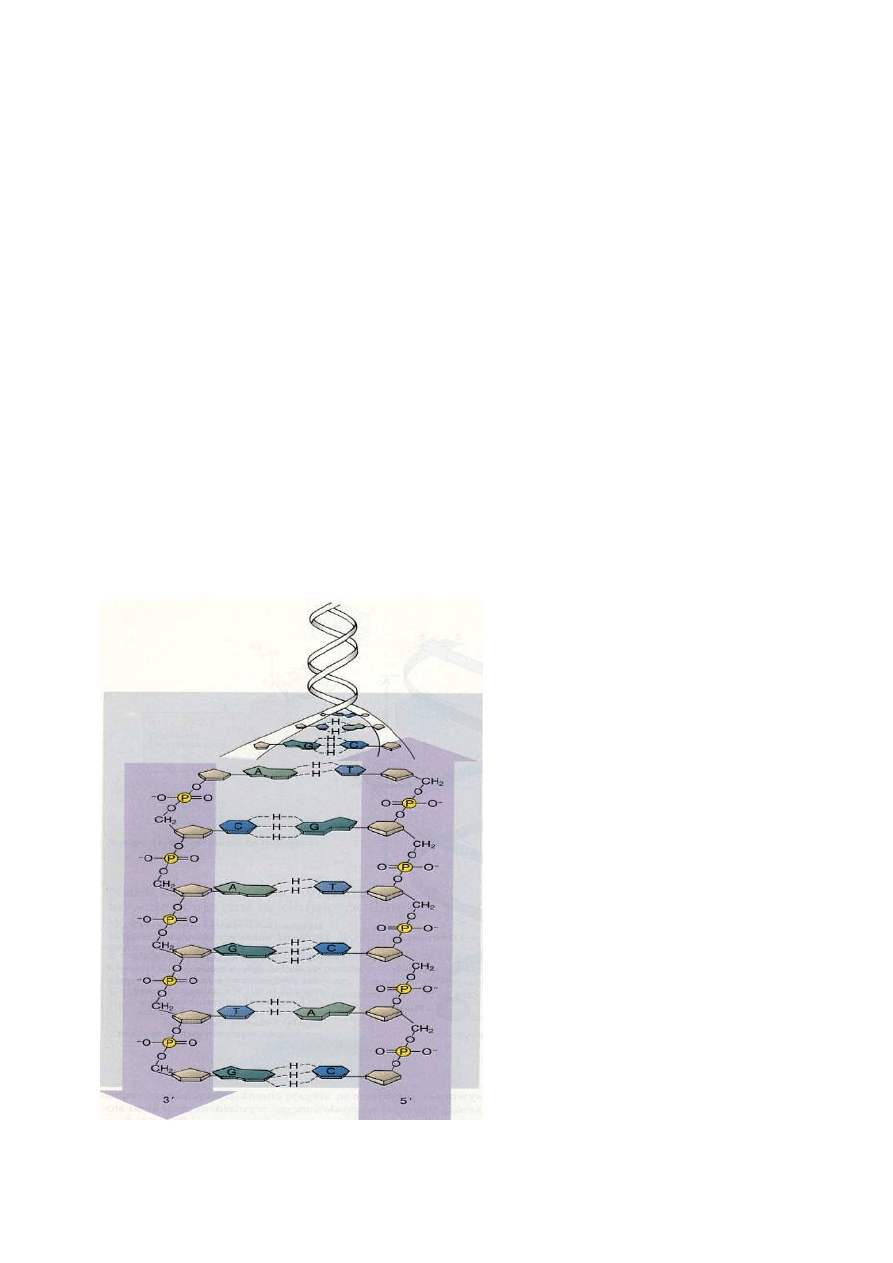
Dalsze prace nad modelem doprowadziły do ustalenia, że adenina może łączyć się w parę z
tymina, a guanina z cytozyną, dzięki tworzącym się między nimi wiązaniom wodorowym;
połączenie między cytozyną a adeniną i guanina a tyminą prowadzi do powstania korzystnego
układu wiązań wodorowych.
Sposób
połączenia
adeniny
z
tyminą i guaniny z cytozyną za pomocą
wiązań wodorowych wygląda następująco:
adeninę z tyminą łączą dwa wiązania
wodorowe, a guaninę z cytozyną - trzy.
Powstawanie specyficznych par A-T i G-C
doskonale tłumaczy regułę Chargaffa.
Liczba cząsteczek cytozyny musi być
równa
liczbie
cząsteczek
guaniny,
ponieważ każdej cząsteczce cytozyny w
jednej nici odpowiada cząsteczka guaniny
w drugiej nici. Analogicznie, każdej
cząsteczce
adeniny
w
jednej
nici
odpowiada cząsteczka tyminy w nici
drugiej. Sekwencje zasad w obu niciach są
zatem
elementarne
(uzupełniają
się
nawzajem), ale nie identyczne. Mówiąc
inaczej, sekwencja nukleotydów w jednej
nici
DNA
wymusza
komplementarną
sekwencję nukleotydów w drugiej nici. Na
przykład, jeśli kolejność nukleotydów w
jednej nici jest:
3' AGCTAC 5'
to druga nić musi mieć komplementarną
sekwencję:
5' TCGATG 3'
Model podwójnego heliksu wyraźnie sugeruje, że w sekwencji zasad w DNA może być zawarta
informacja genetyczna. Ograniczenie dowolności łączenia się zasad w pary w żaden sposób nie
wpływa na możliwość występowania różnorodnych sekwencji, których liczba w łańcuchu
polinukleotydowym jest w zasadzie nieograniczona. Ponieważ cząsteczki DNA występujące w
komórkach zbudowane są z milionów nukleotydów, może być w nich zawarta ogromna ilość
informacji.
Replikacja DNA jest semikonserwatywna: ka
ż
da dwuniciowa cz
ą
steczka DNA zawiera ni
ć
star
ą
oraz nowo zsyntetyzowan
ą
Dwie wyraźnie rzucające się w oczy
cechy modelu DNA stworzonego przez
Watsona i Cricka wskazywały, że DNA
jest najprawdopodobniej materiałem
genetycznym.
Jedna
z
nich,
to
wspomniana
już
możliwość
kodowania
informacji
w
postaci
sekwencji zasad. Drugą, niezwykłą
cechą
modelu
jest
możliwość
precyzyjnego kopiowania zawartej w
sekwencji zasad informacji, czyli
replikacji
DNA.
Biologiczny
sens
replikacji był dla twórców modelu
oczywisty, czego potwierdzeniem było
słynne już stwierdzenie zamieszczone
na końcu ich pierwszego, krótkiego
doniesienia:
"Nie
uszło
naszej
uwadze, że postulowane przez nas
specyficzne
łączenie
się
zasad
sugeruje natychmiast prawdopodobny
mechanizm
kopiowania
materiału
genetycznego".
Z reguły komplementarności
zasad wypływa wniosek, że każda nić
w cząsteczce DNA może służyć jako
matryca do syntezy nici biegnącej w
przeciwnym kierunku. Proces taki
wymagałby
zerwania
wiązań
wodorowych pomiędzy obiema nićmi
w celu ich rozdzielania. Każda z rozdzielonych nici mogłaby dobudowywać na zasadzie
komplementarności kolejne nukleotydy, aż zostałaby odtworzona nić siostrzana. W rezultacie
powstałyby dwie cząsteczki DNA o strukturze podwójnego heliksu, takie same jak cząsteczka
wyjściowa (rodzicielska). Każda z nich miałaby jedną starą nić, pochodzącą z cząsteczki
rodzicielskiej, oraz drugą, nowo zsyntetyzowaną nić komplementarną do starej. Taki sposób
kopiowania informacji zawartej w DNA nosi nazwę replikacji semikonserwatywnej.
Stwierdzenie, że DNA powiela się według opisanego wyżej mechanizmu, umożliwiło wyjaśnienie
trzeciej istotnej cechy materiału genetycznego, jaką jest zdolność do mutowania. Mutacje, czyli
zmiany genetyczne, następują w genach i są wiernie przekazywane potomstwu. Model podwójnego
heliksu skłaniał do przyjęcia koncepcji, zgodnie z którą mutacje polegają na zmianie sekwencji
zasad w DNA. Jeżeli kopiowanie DNA jest wynikiem tworzenia łańcucha komplementarnych zasad,
każda zmiana sekwencji zasad w jednej nici powodowałaby powstanie nowego układu zasad w
kolejnym cyklu replikacyjnym. Nowa sekwencja zasad byłaby przenoszona na cząsteczki potomne
dzięki takiemu samemu mechanizmowi replikacji, jaki został wykorzystany do kopiowania
cząsteczki wyjściowej, działającemu niezmiennie w taki sam sposób, niezależnie od zmiany, jaka
się dokonała w sekwencji zasad. Jakkolwiek semikonserwatywny mechanizm replikacji
zaproponowany przez Watsona i Cricka był (i jest) prosty i przekonujący, wymagał on
potwierdzenia eksperymentalnego. Przede wszystkim trzeba było wykluczyć kilka innych
możliwości. Na przykład zgodnie z konserwatywnym mechanizmem replikacji obie nici
rodzicielskiego
DNA
pozostawałyby stale razem, a
nici potomne tworzyłyby nowy
podwójny heliks. W wyniku
jeszcze
innego
możliwego
sposobu
samopowielania
się
DNA, zwanego dyspersyjnym,
powstawałyby
cząsteczki,
w
których każda z nici byłaby
złożona
z
pewnej
liczby
fragmentów starych nici oraz
pewnej liczby fragmentów nici
nowo
zsyntetyzowanych.
Udowod-nienie
tezy,
że
replikacja przebiega zgodnie z
mechanizmem
semi-
konserwatywnym,
wymagało
znalezienia
sposobu
na
rozróżnienie
nici
starej
od
nowoutworzonej. Stało się to
możliwe dzięki zastosowaniu do
znakowania
nici
DNA
"ciężkiego" izotopu azotu 15N
(w
przyrodzie
powszechnie
występuje
azot
14N).
Do
rozdzielania dużych cząsteczek,
takich jak DNA, wykorzystuje
się
różnicę
ich
gęstości,
stosując
technikę
zwaną
wirowaniem
w
gradiencie
stężeń. Gdy DNA zmiesza się z
roztworem chlorku cezu (CsCl) i
wiruje z dużą szybkością, roztwór soli tworzy w probówce gradient gęstości; w górnej warstwie
gęstość roztworu jest najmniejsza, na dnie probówki zaś największa. W czasie wirowania cząsteczki
DNA przemieszczają się w probówce do takiego miejsca, w którym gęstość roztworu CsCl jest taka
sama jak gęstość DNA.
W 1957 r. Matthew Meselson i Franklin Stahl przeprowadzili pomysłowo zaplanowane
doświadczenie. Hodowali oni bakterie Escherichia coli na pożywce zawierającej izotop 15N w
postaci chlorku amonowego (NH4Cl). Bakterie wykorzystywały "ciężki azot" do syntezy zasad
azotowych, które następnie włączane były do DNA. Powstałe w ten sposób cząsteczki DNA,
zawierające izotop 15N, wydzielano z pewnej części bakterii; po wirowaniu w gradiencie stężenia
chlorku cezu cząsteczki ciężkiego DNA lokowały się blisko dna probówki. Pozostałe bakterie (które
również zawierały DNA znaczony azotem 15N) po przeniesieniu na pożywkę z chlorkiem
amonowym zawierającym powszechnie występujący lżejszy izotop 14N przechodziły kilka cykli
podziałowych.
Oczekiwano, że nowo zsyntetyzowane nici DNA będą lżejsze, ponieważ do ich budowy bakterie
użyły lekkiego izotopu 14N. Istotnie, cząsteczki DNA wyizolowane z bakterii powstałych po
pierwszym podziale komórkowym miały gęstość, która wskazywała, że zawierają one o połowę
mniej izotopu 15N niż cząsteczki DNA wyjściowego (całkowicie "ciężkiego DNA"). W następnym
cyklu podziałowym pojawiły się dwa rodzaje cząsteczek DNA: jedne o gęstości pośredniej,
wskazującej, że są to cząsteczki-hybrydy wyznakowane w równym stopniu oboma izotopami azotu
(14N i 15N), oraz drugie, zawierające wyłącznie lekki izotop 14N. Każda zatem nić wyjściowego
podwójnego heliksu (DNA rodzicielskiego) stała się częścią składową innej potomnej cząsteczki
DNA, dokładnie tak jak przewidywał to model semikonserwatywnej replikacji.
Replikacja DNA jest zło
ż
onym procesem wyró
ż
niaj
ą
cym si
ę
wieloma szczególnymi cechami
Ogólne zasady replikacji DNA wynikają w oczywisty sposób z modelu zaproponowanego przez
Watsona i Cricka, jednak do przebiegu procesu niezbędny jest udział złożonego układu licznych
białek i enzymów, których precyzyjne współdziałanie kojarzyć się może z pracą skomplikowanej
maszyny. Wiele zasadniczych cech tego procesu ma charakter uniwersalny. Niezależnie od tego,
pewne cechy replikacji są różne u prokariontów i eukariontów, co wynika z odmiennej organizacji
przestrzennej ich materiału genetycznego. W komórkach bakterii, takich jak E. coli, większość lub
cały materiał genetyczny występuje w postaci pojedynczej kolistej cząsteczki dwuniciowego DNA.
Każdy nie zreplikowany chromosom eukariotyczny zawiera pojedynczą, liniową cząsteczkę
dwuniciowego DNA zasocjowaną z wielką liczbą cząsteczek białek i RNA.
Podczas replikacji dwuniciowy DNA musi zosta
ć
rozpleciony
Watson i Crick zdawali sobie
sprawę
z
tego,
że
w
stworzonym
przez
nich
modelu podwójnego heliksu
obie nici muszą być wokół
siebie oplecione, podobnie
jak sznurki w linie. Gdy
usiłujemy
rozdzielić
te
sznurki, w linie powstają
napięcia, na skutek których
okręca
się
ona
wokół
własnej osi albo skręca się
w jeszcze ściślejsze zwoje.
Można
by
oczekiwać
podobnego zachowania się
DNA podczas rozplatania
jego dwóch nici. Tak się
jednak
nie
dzieje.
Rozplatanie dokonuje się
przy
udziale
enzymu
zwanego
helikazą
DNA,
która
przesuwając
się
wzdłuż podwójnego heliksu
ułatwia
rozkręcanie
obu
nici.
Gdy
nici
są
już
rozdzielone, przyłączają się
do
nich
białka
destabilizujące
heliks,
zapobiegające
jego
odtwarzaniu się, zanim nie
nastąpi
skopiowanie
obu
nici. Ponieważ cząsteczki
DNA są bardzo długie i
cienkie,
tworzące
się
podczas
ich
rozplatania
napięcia muszą zostać w jakiś sposób zlikwidowane. Dokonują tego specyficzne enzymy zwane
topoizomerazami, które przecinają cząsteczkę DNA w miejscu naprężenia, a potem ponownie ją
łączą, efektywnie "rozsupłując" w ten sposób węzły powstające podczas replikacji.
DNA syntetyzowany jest zawsze w kierunku od 5' do 3'
Reakcję łączenia nukleotydów w łańcuch polinukleotydowy katalizują enzymy zwane polimerazami
DNA. Działanie tych enzymów jest ograniczone pewnymi regułami, co stanowi jedną z przyczyn
złożoności procesu replikacji. Polimerazy DNA mogą dołączać nowe nukleotydy wyłącznie do końca
3' nowo syntetyzowanego łańcucha, komplementarnego do nici kopiowanej .Substratami tej reakcji
są trifosforany nukleozydów; wszystkie one, podobnie jak ATP, zawierają w cząsteczce trzy reszty
fosforanowe dołączone do węgla 5' cukru oraz zasadę azotową. Połączeniu nukleotydów towarzyszy
uwolnienie dwóch reszt fosforanowych. Reakcja ta, podobnie jak reakcja hydrolizy ATP, jest silnie
egzergoniczna i w związku z tym nie wymaga dostarczania dodatkowej energii. Wydłużanie się
(elongacja) nowej nici polinukleotydowej polega na dołączaniu kolejnych jednostek nukleotydowych
do węgla w pozycji 3' cząsteczki cukru występującego na końcu nici, dlatego nowa nić powstaje
zawsze w kierunku od 5' do 3'.

Do rozpocz
ę
cia syntezy DNA potrzebny jest starter
Drugim ograniczeniem w działaniu polimeraz DNA jest to, że mogą one dobudowywać nukleotydy
tylko do końca 3' istniejącego już fragmentu nici polinukleotydowej. Jak zatem może się odbywać
synteza DNA na rozdzielonych niciach? Możliwość taką stwarza synteza krótkiego (złożonego
zwykle z około pięciu nukleotydów) starterowego RNA, nazywanego często po prostu starterem lub
na wzór nazwy angielskiej primerem, syntetyzowanego przez białkowy kompleks zwany
primosomem. Starterowy RNA jest polimerem rybonukleotydów, czyli kwasem rybonukleinowym,
który łączy się z jednoniciową matrycą DNA, zgodnie z regułą komplementarności zasad, w
miejscu, w którym rozpoczyna się proces replikacji. Polimeraza DNA może teraz dobudowywać
nowe podjednostki nukleotydowe do końca 3' starterowego RNA, który zostanie wkrótce wycięty i
rozłożony, a jego miejsce wypełni fragment DNA.
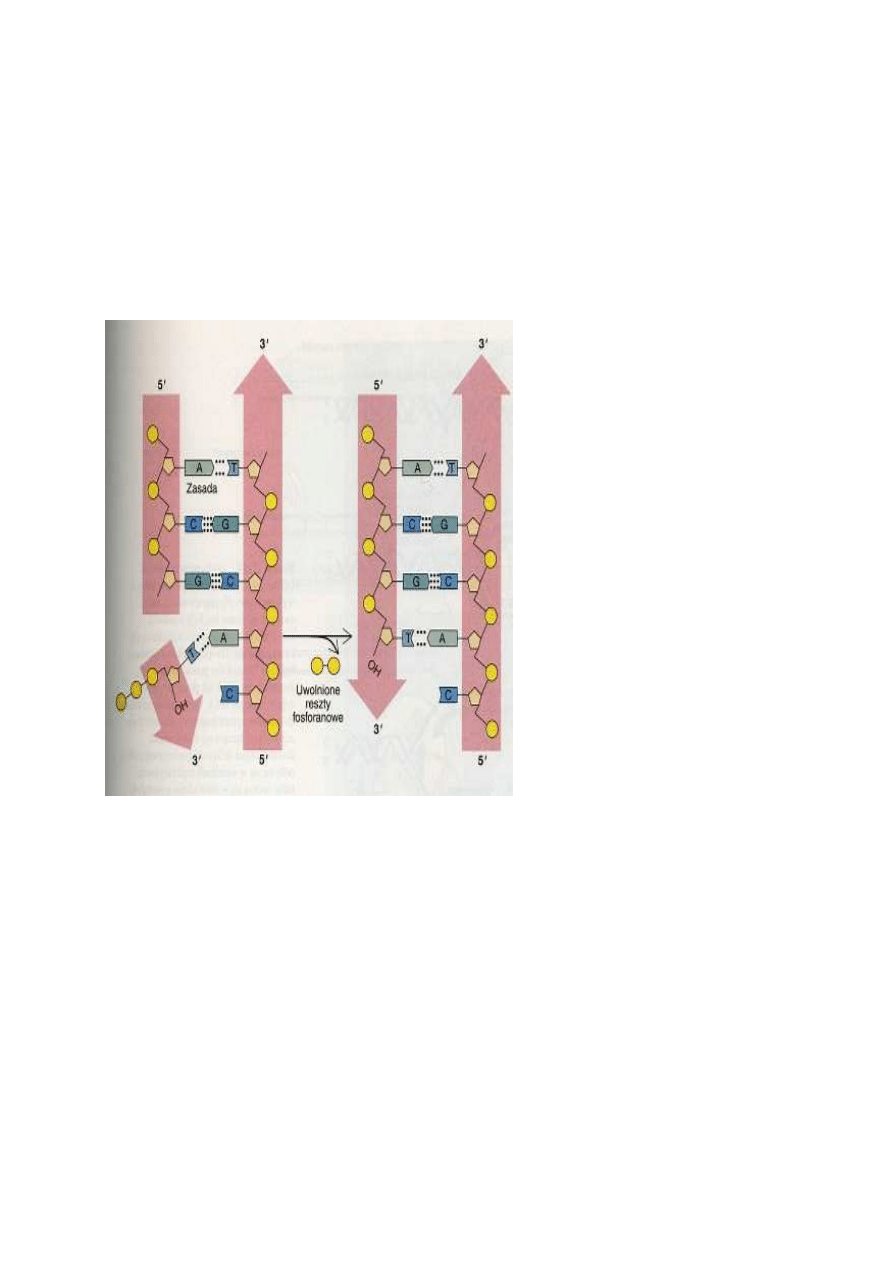
Replikacja jednej nici przebiega w sposób ci
ą
gły, replikacja drugiej odbywa si
ę
fragmentami
Poważną przeszkodę w zrozumieniu mechanizmu replikacji stanowił fakt, że komplementarne nici
DNA ułożone są w przeciwnych kierunkach. W związku z tym, że synteza przebiega wyłącznie w
kierunku od 5' do 3' (co oznacza, że odczytywanie matrycy odbywa się w kierunku od 3' do 5'),
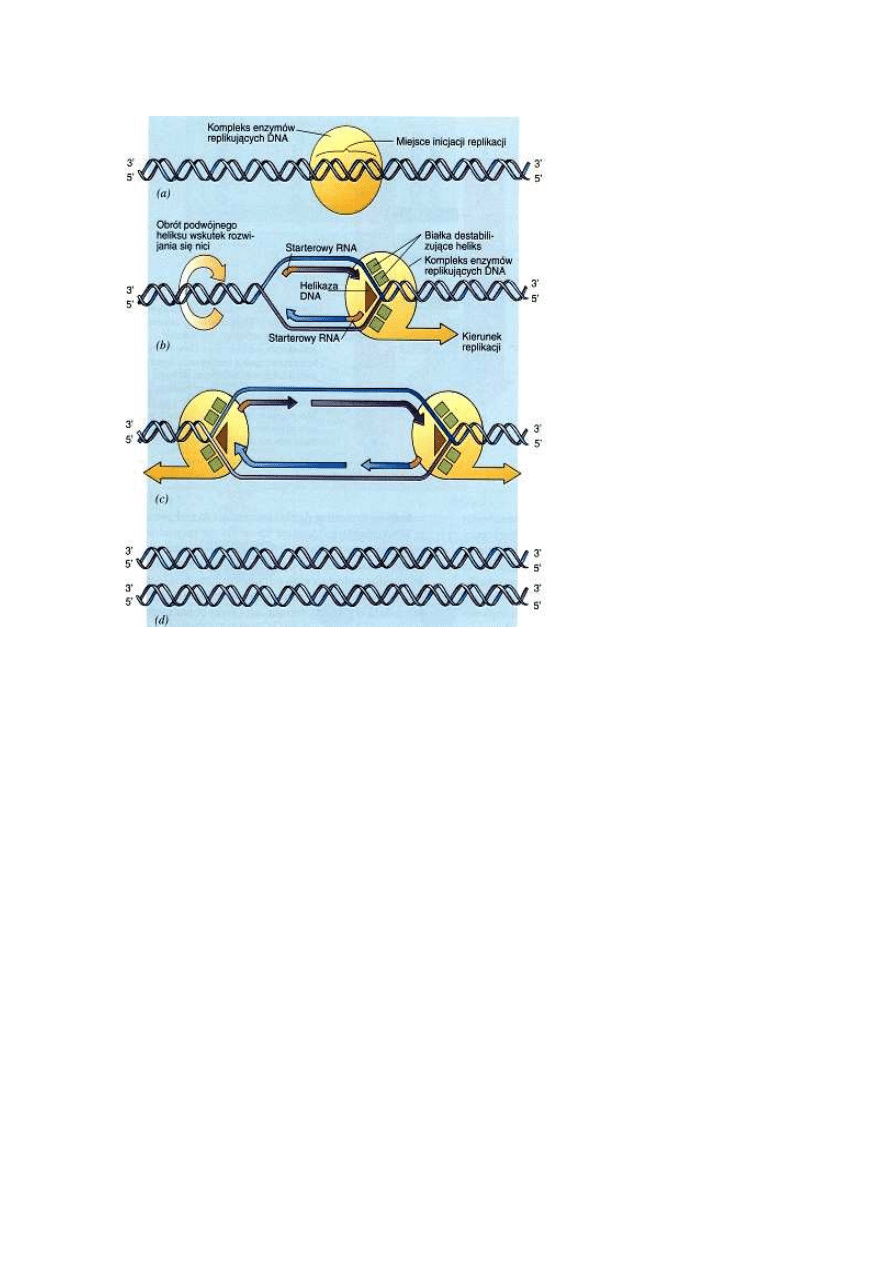
kopiowanie każdej z nici musiałoby się zaczynać na przeciwnych końcach podwójnego heliksu. Tak
się jednak nie dzieje. Replikacja DNA rozpoczyna się w specyficznym miejscu cząsteczek DNA,
zwanym miejscem inicjacji replikacji, i przebiega jednocześnie na obu niciach w obszarze noszącym
nazwę widełek replikacyjnych. Wydłużanie jednej z powstających nici odbywa się zgodnie z ruchem
widełek replikacyjnych, przebiega zatem w sposób ciągły. Nić tę określa się mianem nici
prowadzącej. Synteza drugiej nici przebiega w kierunku przeciwnym do ruchu widełek
replikacyjnych. Powstaje ona w postaci krótkich (zawierających od 100 do 1000 nukleotydów)
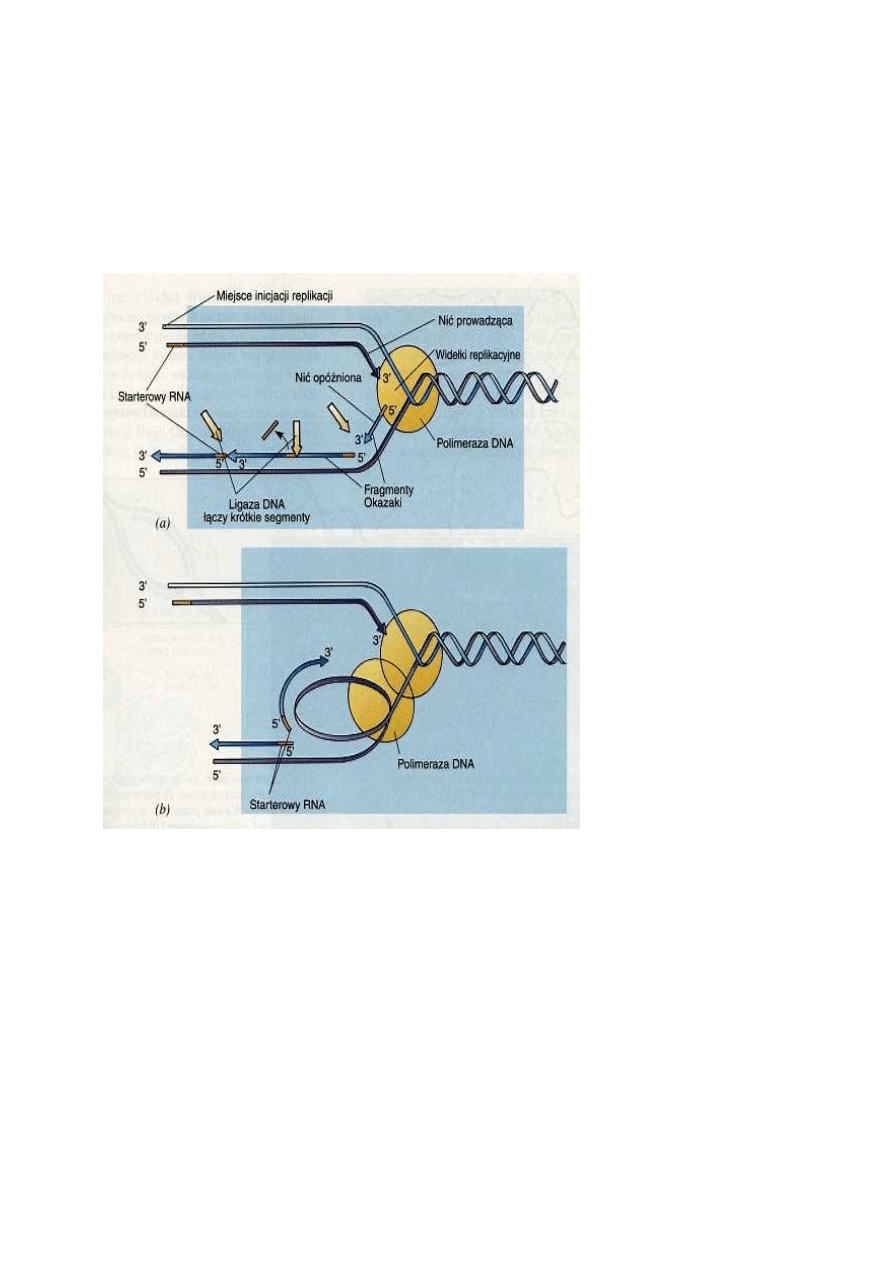
fragmentów Okazaki, nazwanych tak dla upamiętnienia ich odkrywcy, Reijii Okazaki. Nić ta nosi
nazwę nici opóźnionej. Syntezę każdego fragmentu Okazaki inicjuje odrębny starterowy RNA;
synteza ta przebiega w kierunku końca 5' poprzednio utworzonego fragmentu. W procesie tym
uczestniczą polimerazy DNA kilku typów, z których każda jest złożonym enzymem
wielofunkcyjnym. Gdy tworzący się właśnie fragment Okazaki zbliża się do fragmentu
zsyntetyzowanego przed nim, jedna z części polimerazy DNA degraduje poprzedni starterowy RNA,
umożliwiając innej polimerazie wypełnienie przerwy między dwoma odcinkami nici DNA.
Ostatecznie oba fragmenty zostają spojone przez ligazę DNA, enzym katalizujący reakcję
zespolenia końca 3' jednego fragmentu DNA z końcem 5' drugiego. Przypuszcza się, że synteza obu
nici, prowadzącej i opóźnionej, odbywa się jednocześnie dzięki temu, że nić opóźniona tworzy
pętlę, umożliwiając w ten sposób dołączanie nowych jednostek nukleotydowych do końców 3'
każdej z nowo syntetyzowanych nici przez pozostającą w widełkach replikacyjnych polimerazę DNA.
Synteza DNA przebiega na ogół w dwóch kierunkach
W wyniku rozdzielenia się obu nici podwójnego heliksu widełki replikacyjne powstają w dwóch
miejscach, tak że poczynając od miejsca inicjacji proces replikacji przebiega jednocześnie w dwóch
kierunkach. W komórkach prokariotycznych inicjacja replikacji następuje zazwyczaj w jednym
punkcie kolistego chromosomu, w związku z czym dwie pary widełek replikacyjnych przesuwają się
wokół kolistej cząsteczki DNA i w końcu spotykają po przeciwnej stronie koła, gdzie następuje
zakończenie procesu syntezy. Chromosom eukariotyczny utworzony jest z jednej bardzo długiej
cząsteczki DNA, której replikację przyspiesza inicjowanie syntezy nowych nici jednocześnie w wielu
miejscach. W każdych widełkach replikacyjnych synteza przebiega aż do momentu zetknięcia się z
widełkami przesuwającymi się w przeciwnym kierunku. Po ukończeniu syntezy przez wszystkie
widełki replikacyjne powstaje chromosom składający się z dwóch cząsteczek dwuniciowego DNA.
DNA w chromosomach upakowany jest w zorganizowane struktury wy
ż
szego rz
ę
du
Komórki prokariotyczne i eukariotyczne różnią się
w istotny sposób zarówno zawartością DNA, jak i
organizacją jego cząsteczek. Pojedynczy kolisty
chromosom bakterii E. coli zawiera DNA złożony z
4 x 106 par zasad (o długości około l ,35 mm).
Długość cząsteczki DNA jest około 1000 razy
większa niż długość samej bakterii. Cząsteczka
DNA musi zatem być ściśle zwinięta i upakowana,
aby zmieścić się w komórce. W utrzymywaniu
zwartej struktury cząsteczki DNA uczestniczą
specjalne białka. Typowa komórka eukariotyczna
zawiera znacznie więcej DNA niż bakteria. DNA
występuje w jądrze w postaci chromosomów,
których liczba i kształt są różne u poszczególnych
gatunków. Jądro komórki ludzkiej ma wymiary
zbliżone do wymiarów bakterii, zawiera jednak
ponad 1000 razy więcej DNA niż E. coli.
Haploidalna zawartość DNA w komórce ludzkiej
wynosi
około
3
x
109
par
zasad;
po
wyprostowaniu ten DNA miałby długość blisko l
m. W komórkach eukariotycznych DNA, którego
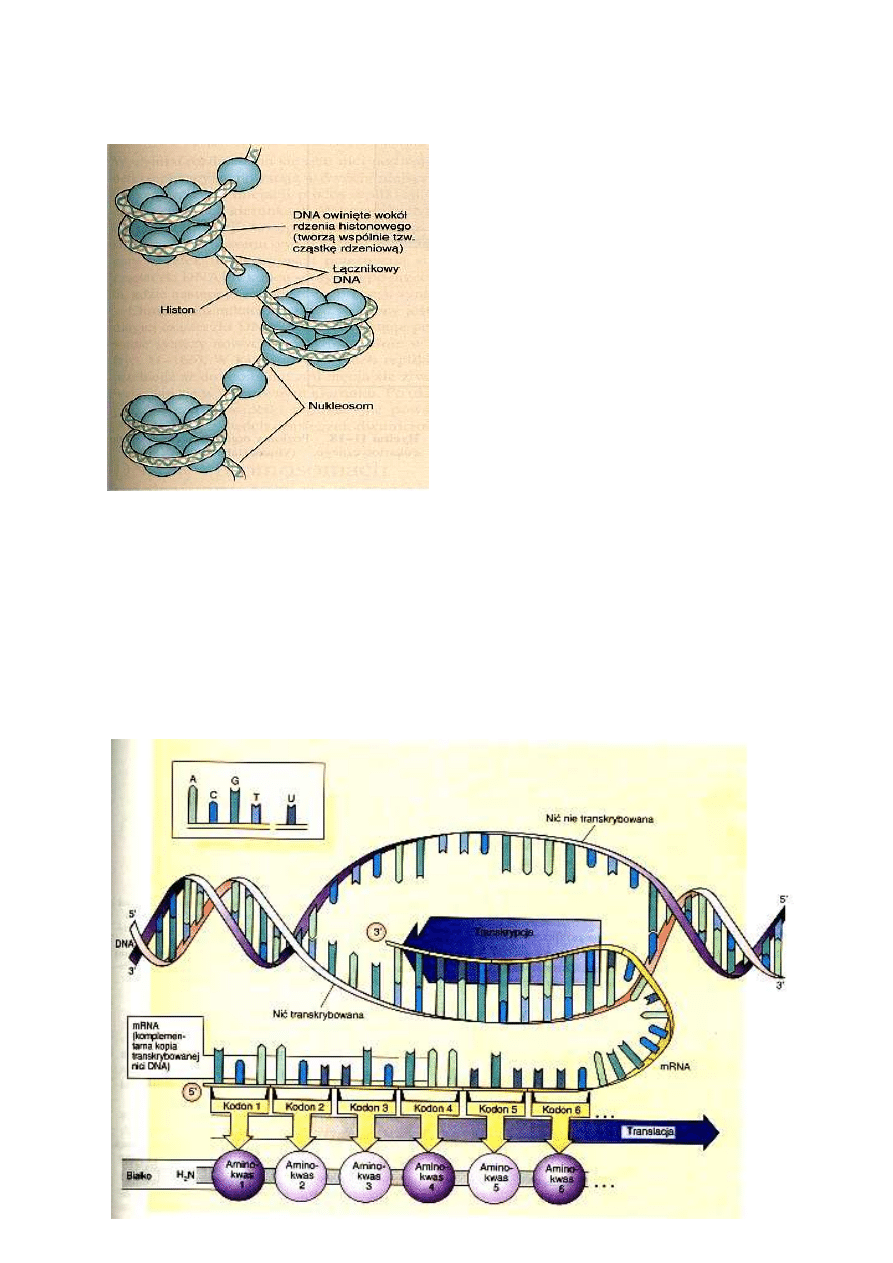
cząsteczka ma kwaśny charakter i ujemny
ładunek, oplata się wokół zasadowych (dodatnio naładowanych) cząsteczek białek histonowych,
tworząc struktury zwane nukleosomami. Podstawowym elementem każdego nukleosomu jest
kulistego kształtu cząstka rdzeniowa, złożona z łańcucha DNA o długości około 140 par zasad
owiniętego wokół dyskowatego rdzenia, zbudowanego z ośmiu cząsteczek histonów. Cząsteczka
innego rodzaju histonu związana jest z DNA łączącym dwie sąsiadujące cząstki rdzeniowe. Włókno
złożone z nukle-osomów jest podstawowym elementem strukturalnym chromatyny, kompleksu
nukleoproteinowego, z którego zbudowane są chromosomy. W chromatynie włókno nukleosomowe
(chromatynowe), skręcone w ścisłą spiralę, tworzy wielkie pętle utrzymywane razem przez zestaw
białek niehistonowych, noszących nazwę białek macierzy jądrowej.
Sekwencja zasad w DNA przepisywana jest na sekwencj
ę
zasad w RNA, która tłumaczona jest
na sekwencj
ę
aminokwasów w białkach

Mimo iż sekwencja zasad w DNA określa sekwencję aminokwasów w białkach, informacja zawarta
w DNA nie jest wykorzystywana bezpośrednio. Jako pośrednik między DNA a białkiem służy inny
kwas nukleinowy, RNA, czyli kwas rybonukleinowy. Podobnie jak DNA, RNA jest polimerem
nukleotydów. Między cząsteczkami obu kwasów nukleinowych występują jednak istotne różnice.
Cząsteczki RNA są zwykle jednoniciowe, choć w ich wewnętrznych obszarach mogą występować
sekwencje komplementarne, które po złożeniu się, tworzą krótkie odcinki dwuniciowe. W RNA
występuje cukier ryboza (a nie deoksyryboza), tyminę zaś zastępuje inna zasada, uracyl. Podobnie
jak tymina, uracyl jest pirymidyną i może tworzyć dwa wiązania wodorowe z adeniną. Uracyl i
adenina tworzą zatem parę komplementarną. W wyniku ekspresji genu kodującego białko
wytwarzana jest cząsteczka RNA stanowiąca kopię informacji zawartej w DNA. Proces ten
przypomina replikację DNA, gdyż sekwencja zasad w nici RNA określana jest przez tworzenie
komplementarnych par z zasadami w jednej z nici DNA. Ponieważ synteza RNA polega na
kopiowaniu informacji zawartej w jednym kwasie nukleinowym (DNA) na informację zawartą w
innym kwasie nukleinowym (RNA), nazywamy ten proces transkrypcją (przepisywaniem). RNA,
który niesie specyficzną informację o budowie białka, nazywany jest matrycowym (lub
informacyjnym) RNA, w skrócie mRNA. W drugim etapie ekspresji genu według informacji zawartej
w mRNA syntezowane jest białko o określonej sekwencji aminokwasów. Proces ten nazywany jest
translacją (tłumaczeniem), ponieważ polega na przetworzeniu "języka kwasów nukleinowych" w
cząsteczce mRNA na "język aminokwasów" w cząsteczce białka. Informacja o budowie białka
zawarta w mRNA zapisana jest za pomocą kodonów, tj. kombinacji trzech następujących po sobie
zasad. Każdy kodon w mRNA określa jeden aminokwas, np. jednym z kodonów, określających
aminokwas treoninę, jest 5'-ACG-3'. Translacja wymaga udziału maszynerii komórkowej, która
potrafi rozpoznać i odszyfrować kodony w mRNA. Kluczowym elementem tej maszynerii są
cząsteczki transportującego (przenośnikowego) RNA (tRNA). Każda cząsteczka tRNA jest
"adapterem", który potrafi połączyć się ze specyficznym aminokwasem i rozpoznać właściwy dla
tego aminokwasu kodon w mRNA. Rozpoznawanie kodonów możliwe jest dzięki obecności w każdej
cząsteczce tRNA sekwencji trzech zasad, zwanej antykodonem, która łączy się z kodonem w mRNA,
tworząc komplementarne pary zasad. W naszym przykładzie dokładnym antykodonem dla treoniny
jest trójka 3'-UGC-5'. Translacja wymaga również połączenia aminokwasów we właściwym
porządku. Dokonują tego rybosomy , złożone organelle utworzone z dwóch różnych podjednostek,
których każda zawiera wiele białek i rybosomowy RNA (rRNA). Rybosomy przyłączają się do końca
cząsteczki mRNA i wędrują wzdłuż niej, umożliwiając cząsteczkom tRNA odszyfrowanie informacji i
odpowiednie ułożenie aminokwasów, które zostają następnie połączone we właściwym porządku
wiązaniami peptydowymi; tak powstaje polipeptyd.Procesy te nie mogłyby zachodzić bez instrukcji
zapisanej kodem genetycznym. Przed odkryciem Watsona i Cricka zagadką było, w jaki sposób
cztery zasady w DNA mogą być wykorzystane do łączenia 20 rodzajów aminokwasów w cząsteczki
białek, których różnorodność w komórce jest ogromna. Gdy uczeni zaczęli rozpatrywać ten
problem, mając przed oczyma nowy model struktury DNA, stwierdzili, że zasady w DNA mogą
służyć jako czteroliterowy alfabet. Trzyliterowe kombinacje czterech zasad (43) umożliwiają
utworzenie 64 "słów", co aż nadto wystarcza do zakodowania wszystkich występujących naturalnie
aminokwasów. Ponad 10 lat później dokonano ostatecznego "złamania" kodu genetycznego.
Potwierdzono istnienie powszechnego we wszystkich organizmach kodu trójkowego, w którym
każde "słowo" składa się z trzech zasad.
Na matrycy DNA transkrybowany jest RNA kilku rodzajów. Znajduje się wśród nich
rybosomowy RNA (rRNA), transportujący RNA (tRNA), a także matrycowy RNA (mRNA). Cząsteczki
RNA syntetyzowane są przez zależne od DNA polimerazy RNA, enzymy występujące we wszystkich
komórkach. Polimerazy te wymagają jako matrycy DNA i przypominają pod wieloma względami
polimerazy DNA. Używają jako substratów trifosforanów nukleozydów (nukleotydów z trzema
resztami fosforanowymi), z których usuwają dwie reszty fosforanowe podczas przyłączania
podjednostek wiązaniem kowalencyjnym do końca 3' RNA.
Sekwencja zasad w matrycowym RNA koduje białko
W rejonie DNA kodującym białko zwykle tylko jedna z nici jest transkrybowana. Rozpatrzmy
fragment nici DNA zwierającej sekwencję zasad 5' - ATTGCCAGA- 3'. Komplementarna do niego nić
ma zapis: 3'- TAACGGTCT - 5', co oznacza zupełnie inną sekwencję aminokwasową. Zatem tylko
jedna z dwóch nici DNA stanowiących gen jest komplementarna do mRNA i to właśnie ta nić jest
transkrybowana. Nazywana jest ona także nicią matrycową. Gdy cząsteczki kwasów nukleinowych
oddziałują z sobą za pomocą komplementarnych par zasad, dwie łączące się nici mają zawsze
przebieg antyrównoległy. Przeciwbieżne w stosunku do siebie są również transkrybowana nić DNA i
komplementarna do niej nić RNA. Cząsteczka dwuniciowego DNA stanowiąca chromosom zawiera
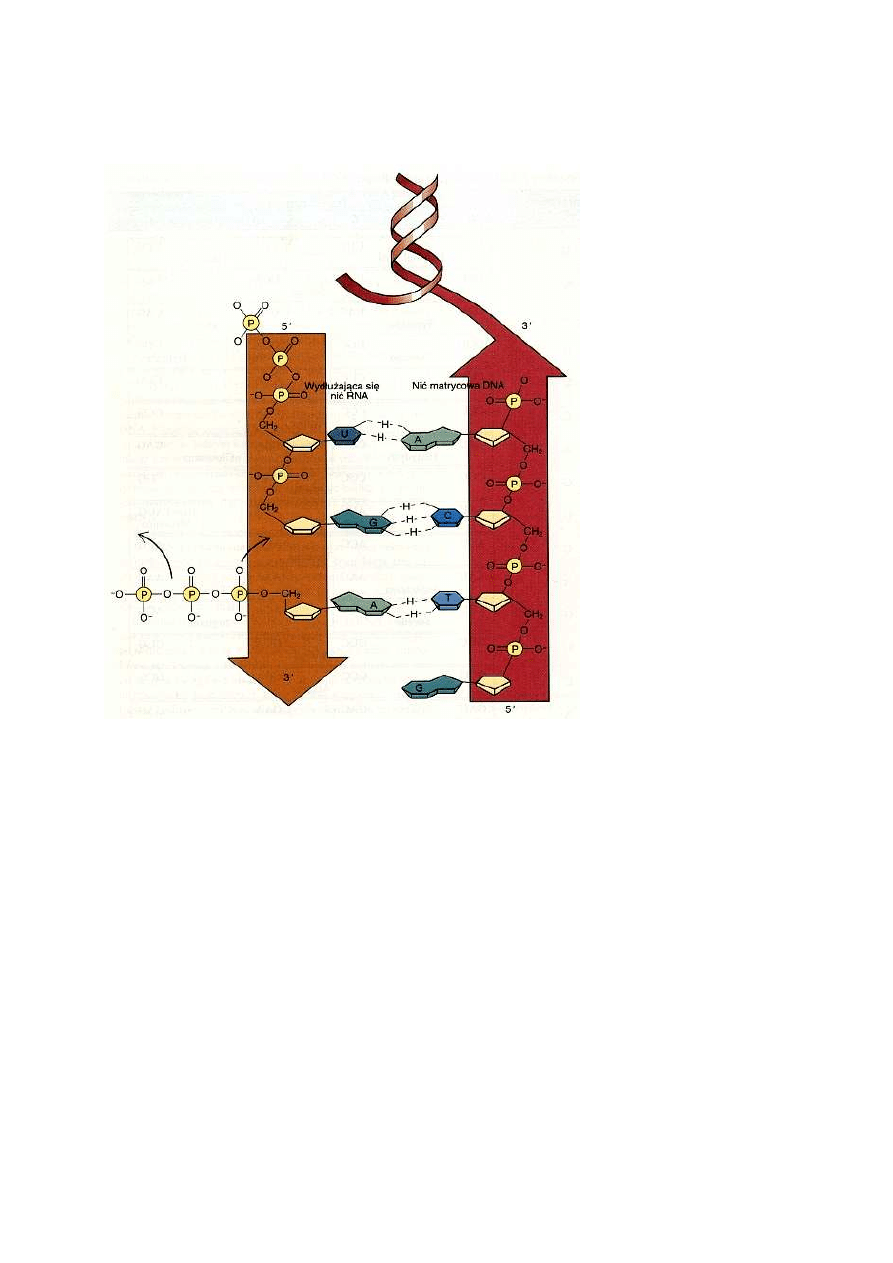
tysiące genów; ta sama nić może być w jednych genach nicią transkrybowana, w innych zaś - nie
transkrybowana .
Proces
transkrypcji
rozpoczyna
się
od
rozpoznania
przez
polimerazę
RNA
specyficznej
sekwencji
zasad,
zwanej
rejonem
promotorowym
lub
promotorem,
znajdującej
się
zwykle
tuż
przed
początkiem
właściwego
genu. W odróżnieniu od
syntezy DNA, synteza RNA
nie
wymaga
startera.
Nukleotyd
na
końcu
5'
zachowuje
swą
grupę
trifosforanową,
ale
z
kolejnych
trifosforanów
nukleotydów przyłączanych
do
końca
3'
rosnącego
łańcucha
dwie
reszty
fosforanowe są usuwane,
pozostała
zaś
staje
się
częścią
szkieletu
fosforanowo-cukrowego
(podobnie
jak
to
ma
miejsce w czasie syntezy
DNA).
Ostatni
z
przyłączonych nukleotydów
ma na końcu 3' wolną grupę
wodorotlenową
.
O sekwencji zasad w genie
lub transkrybowanym na
nim mRNA mówimy, że leży
"powyżej" (ang. upstream)
lub
"poniief
(ang.
downstream) w stosunku do przyjętego miejsca odniesienia. Kryterium jest tu kierunek
transkrypcji. Powyżej danego miejsca znajdują się sekwencje położone w kierunku końca 5'
sekwencji mRNA lub końca 3' transkrybowanej nici DNA (przeciwnie do kierunku transkrypcji).
Poniżej danego miejsca znajdują się sekwencje położone w kierunku końca 3' cząsteczki RNA lub
końca 5' transkrybowanej nici DNA (zgodnie z kierunkiem transkrypcji).
"Powyżej" "Poniżej"
5'-A-T-G-A-C-T-3' (nie transkrybowana nić DNA) 3'-T-A-C-T-G-C-5' (transkrybowana nić DNA)
Kierunek transkrypcji->
Trifosforan 5'-A-U-G-A-C-U-3' OH (RNA)
W bakterii Escherichia coli transkrypcja genu zostaje zainicjowana, gdy polimeraza RNA (z udziałem
innego białka) rozpozna specyficzną promotorową sekwencję zasad leżącą powyżej sekwencji
kodującej
białko.
Różne geny mogą mieć nieco różne sekwencje promotorowe, komórka może więc "decydować",
które geny mają być w określonym momencie transkrybowane. Promotory bakteryjne liczą na ogół
około 40 zasad i w DNA znajdują się tuż powyżej miejsca, od którego rozpoczyna się transkrypcja.
Z chwilą gdy polimeraza rozpozna właściwy promotor, dokonuje ona rozplecenia podwójngo heliksu
DNA
i
rozpoczyna
transkrypcję
nici,
która
stanowi
matrycę.
O zakończeniu (terminacji) transkrypcji, podobnie jak o jej rozpoczęciu (inicjacji), decyduje zestaw
specyficznych sekwencji zasad. Te znajdujące się na końcu genu sekwencje działają jak sygnał
"stop" dla polimerazy RNA.
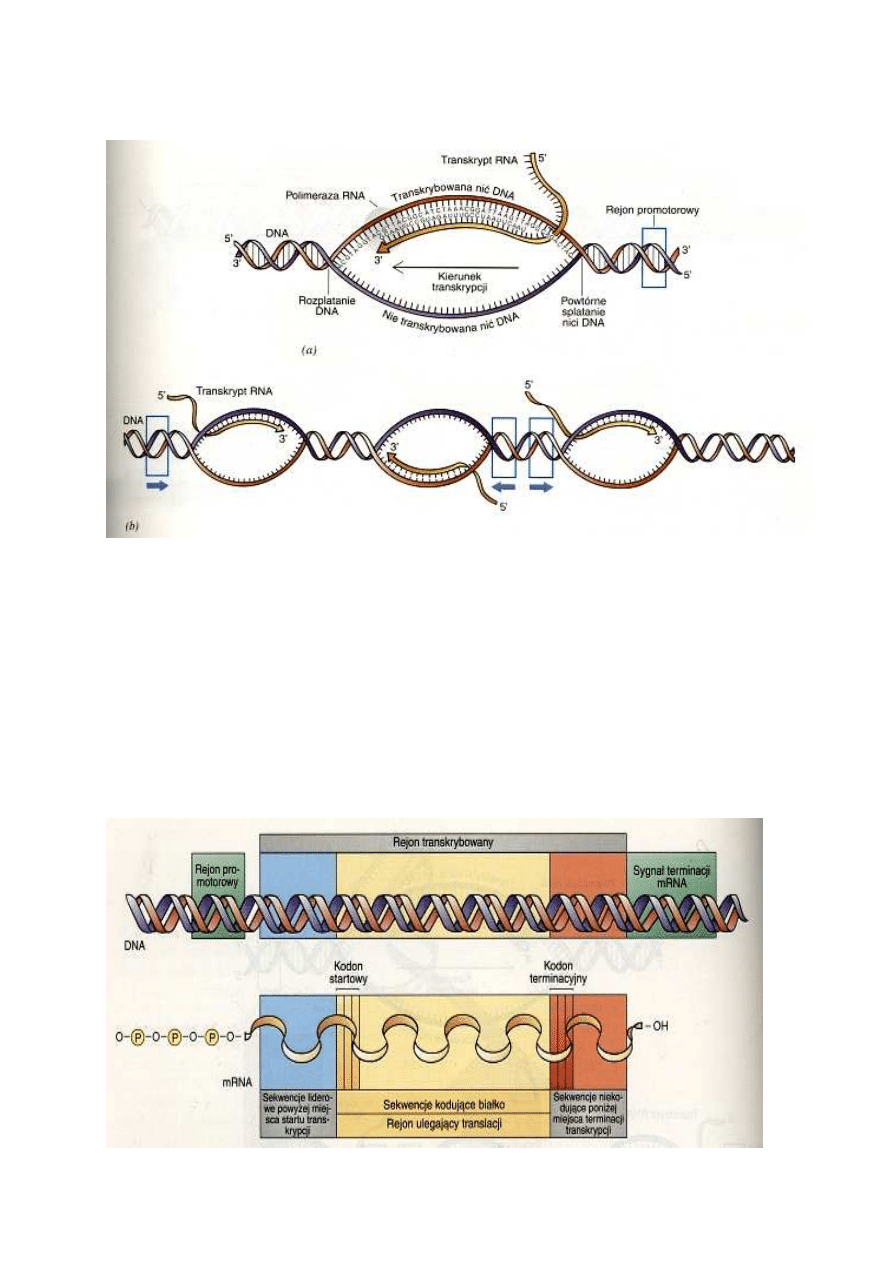
Matrycowy RNA zawiera dodatkowe sekwencje zasad, które nie koduj
ą
bezpo
ś
rednio białka
Całkowity bakteryjny mRNA, obok sekwencji zasad kodującej białko, zawiera sekwencje
dodatkowe. Polimeraza RNA rozpoczyna transkrypcję genu od miejsca leżącego znacznie powyżej
sekwencji kodującej białko. W rezultacie, mRNA ma na końcu 5' sekwencję niekodującą, którą
nazywamy sekwencją liderową. W sekwencji liderowej znajdują się sygnały rozpoznawcze dla
rybosomu, które umożliwiają właściwe umiejscowienie na mRNA rybosomów dokonujących jego
translacji. W komórkach bakteryjnych w jednej cząsteczce mRNA może być zakodowane jedno
białko lub większa liczba białek. Za sekwencją liderową rozpoczyna się sekwencja kodująca,
zawierająca właściwą informację o budowie białka. Na końcu sekwencji kodującej znajdują się
specjalne sygnały terminacyjne, które wyznaczają koniec cząsteczki białka. Po nich następują
jeszcze 3'- końcowe sekwencje niekodujące, które w różnych mRNA mogą być różnej długości.

Gen definiowany jest jako jednostka funkcjonalna
W swoim czasie wygodnie było definiować gen jako sekwencję nukleotydów kodującą jeden
łańcuch polipeptydowy. Nowe informacje o funkcjonowaniu genów zmusiły uczonych do
rewizji tej definicji. Wiemy dziś, że pewne geny służą wyłącznie do kodowania cząsteczek
RNA, takich jak rRNA i tRNA lub RNA wchodzący w skład małych jądrowych kompleksów
rybonukleoproteinowych, biorących udział w modyfikowaniu złożonych cząsteczek mRNA.
Ostatnie badania wykazały też, że w komórkach eukariotycznych pojedynczy gen, dzięki
zróżnicowanym sposobom modyfikacji mRNA, może służyć do wytwarzania więcej niż
jednego łańcucha polipeptydowego. Gen można zdefiniować, biorąc pod uwagę jego
ostateczny produkt. Jedna z użytecznych definicji stwierdza, że gen zawiera transkrybowaną
sekwencją nukleotydową (oraz towarzyszce sekwencje regulujące jej transkrypcję), która
koduje produkt pełniący specyficzną funkcję w komórce.
Wyszukiwarka
Podobne podstrony:
więcej podobnych podstron