Brakuje mi kilku rzeczy. Poza tym ok.
Ocena: 4,5
Wstęp teoretyczny
BRENDA
Jest to baza danych zawierająca informacje dotyczące białek, ale w szczególności enzymów. Enzymy klasyfikowane są zgodnie z klasyfikacją Komisji Enzymatycznej. Baza uaktualniana jest dwa razy w roku. Informacje pochodzą literatury i czasopism naukowych Baza zwiera min. następujące dan:
nomenklatura enzymu (np. numer EC, synonimy, nazwa systematyczna)
oddziaływania enzym - ligand (np. substraty, produkty, kofaktory, aktywatory, inhibitory)
parametry opisujące enzym (np. wartość KM, optymalne pH i temperatura)
informacje na temat organizmów, z których pochodzi enzym (organizm, lokalizacja, tkanka, z której został wyizolowany)
struktura enzymu (np. sekwencja, masa cząsteczkowa, podjednostki)
właściwości (np. termostabilność, stabilność w różnych pH, zastosowanie).
Brenda posaida także linki łączące z innymi bazami danych, np. z literaturową bazą danych PubMed, bazą KEGG, ExPASY, NCBI, PDB, CATH, UniProt.
KEGG
Jest to baza danych zawierająca informacje na temat genomów, genów, szlaków metabolicznych i metabolitów. Baza KEGG dzieli się na następujące części:, np.:
KEGG 2 - zawiera spis treści bazy KEGG
KEGG Pathway - zawiera mapy szlaków metabolicznych
KEGG Genes - zawiera informacje o genach i białkach
KEGG Disease - zawiera informacja o ludzkich chorobach genetycznych
KEGG Genome - zawiera informacje o genomach
KEGG Orthology - zawiera informacje o grupach ortologów
KEGG Reaction - zawiera informacje o reakcjach biochemicznych
KEGG Ligand - zawiera informacje o związkach i reakcjach chemicznych zachodzących w komórkach
KEGG Organisms - umożliwia wybór szukanego organizmu, łączy głównie z bazą RefSeq
KEGG Medicus - zawiera informacje o chorobach, lekach lub innych substancjach
KEGG Mapper - zawiera narzędzia do tworzenia map szlaków metabolicznych
PUBCHEM
Dzieli się na trze części:
Bioassay - zawiera informacje na temat biologicznej aktywności małych związków chemicznych. Zawiera informacje takie jak np.: numer identyfikacyjny AID, nazwa związku, opis działania, metoda wykorzystana do badania związku
Compound - zawiera obszerną charakterystykę związków chemicznych, min, opis działania, klasyfikację chemiczną, synonimy, masa cząsteczkowa, wzór chemiczny, ładunek, liczba izotopów linki do literaturowej bazy danych PubMed oraz do PubChem Bioassay, Zawiera także linki do związków chemicznych o podobnej budowie, substratach,, informacje o grupie związków do której należy poszukiwany związek, wzór strukturalny z zaznaczonymi grupami hydrofilowymi i hydrofobowymi.
Substance - zawiera podstawowe informacje o związkach chemicznych. Informacje są podobne jak te w bazie PubChem Compound, ale baza ta jest mniej obszerna.
CATH
Zawiera klasyfikację struktur domen białkowych. Białko podzielono na osobne domeny strukturalne i sklasyfikowano według homologicznych nadrodzin. Do klasyfikacji tej kombinację technik automatycznych i manualnych, wykorzystujących algorytmy obliczeniowe, dane empiryczne i statystyczne. Wykorzystano także dane literaturowe oraz wyniki analizy ekspertów. W bazie CATH istnieją cztery główne poziomy hierarchii:
Klasa (Class) - poziom C - wynika ze struktury drugorzędowej białek i upakowania. Obejmuje 4 klasy (alfa-helisa, beta-helisa, alfa/beta, alfa + beta)
Architektura (Architecture) - poziom A - opisuje kształt domeny, wynikający ze struktury drugorzędowej, ale nie wynikający z połączeń tych struktur. Wyróżniamy strukturę typu beczka oraz 3-warstwowa kanapka
Topologia (topology) - poziom T - podział ze względu na kształt domeny i połączenie struktur drugorzędowych w rdzeniu domeny. Wyróżniamy strukturę beta-2-warstwowej kanapki oraz strukturę alfa-beta-3-warstwowej kanapki
Homologiczna nadrodzina (Homologous superfamily) - poziom H - obejmuje organizmy posiadające wspólnego przodka.
SCOP
--> Służy do strukturalnj klasyfikacji białek. Hierarchia SCOP polega na podziale według:
klasy ( alfa-helisa, beta-helisa, alfa/beta, alfa + beta; białka wielodomenowe; małe białka, białka membranowe, białka niezbadane)
sposobu fałdowania
nadrodzin
rodzin[Author:K]
Ćwiczenie 1
Do wyszukania niektórych informacji na temat beta-galaktozydazy pochodzącej z Penicilium chrysogenum wykorzystałam bazę danych BRENDA. Wpisałam w okno wyszukiwania 'beta galactosidase' i wybrałam odpowiedni organizm.
Numer dostępowy poszukiwanej beta-galaktozydazy to EC 3.2.1.23. Beta-galaktozydaza zlokalizowana jest w płynie wewnątrzkomórkowym. Charakteryzują ją następujące parametry:
stała Michaelisa KM = 1,81mM
pH optymalne = 4,0
temperatura optymalna = 30°C
punkt izoelektryczny --> pJ [Author:K] = 4,9.
Enzym ten katalizuje reakcję hydrolizy wiązań o-glikozydowych. Schemat tej reakcji przedstawiony został poniżej.
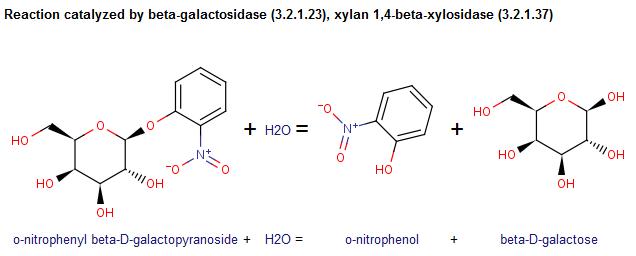
Substratami tej reakcji są o-nitrofenylo-beta-D-galaktopiranozyd oraz woda. Produkty to natomiast o-nitrofenol oraz beta-D-galaktoza. Wzory związków przedstawia powyższa reakcja. Informacje na temat substratów i produktów znalazłam w bazie PubChem Compound. Poniżej przedstawiłam niektóre z informacji.
O-nitrofenylo-beta-D-galaktopiranozyd ma masę cząsteczkową 301.2494 [g/mol]. Jego wzór sumaryczny to C12H15NO8. Może być donorem 4 wodorów oraz akceptorem 8 wodorów. Jego dokładna masa to 301.079766, a masa monoizotopowa to 301.079766. Posiada 21 atomów ciężkich. Jego ładunek wynosi 0. Nie posiada żadnych izotopów. Nie ma żadnych zdefiniowanych atomów posiadających centrum stereogeniczne, ma jednak 5 takich niezdefiniowanych atomów. Kompleksowość wynosi 362.
Beta-D-galaktoza ma masę cząsteczkową 180.15588 [g/mol], a jej wzór sumaryczny to C6H12O6. Jest donorem 5 wodorów i akceptorem 6 wodorów. W cząsteczce może wystąpić jedna rotacja wokół wiązania. Dokładna masa beta-D-galaktozy to 180.063388. Beta-D-galaktoza posiada 12 atomów ciężkich a jej ładunek formalny to 0. Nie ma żadnych izotopów. Ma 5 zdefiniowanych atomów posiadających centrum stereogeniczne, ale żadnych niezdefiniowanych takich atomów. Kompleksowość wynosi 151. W raporcie przedstawiłam jedną z klasyfikacji: węglowodan ->monosacharyd -> heksoza-> galaktoza.
O-nitrofenol ma masę cząsteczkową 139.1088 [g/mol], a jego wzór sumaryczny to C6H5NO3. Jest donorem 1 wodoru, a akceptorem 3 wodorów. W cząsteczce nie występują żadne rotacje wokół wiązań. Dokładna masa tego związku to 139.026943. Masa monoizotopowa to 139.026943. O=nitofenol posiada 10 atomów ciężkich. Jego ładunek formalny to 0. Nie mżadnych izotopów. Nie ma żadnych zdefiniowanych i niezdefiniowanych atomów posioadających centrum stereogeniczne. Kompleksowość wynosi 231. O-nitrofenol sklasyfikowany jest na kilka sposobów, np: poliatomowa jednostka -> heteroatomowa molekularna jednostka->wodorotlenek->związek organiczny z grupą hydroksylową-> fenol-> nitrofenol -> mononitrofenol-> 2-nitrofenol.
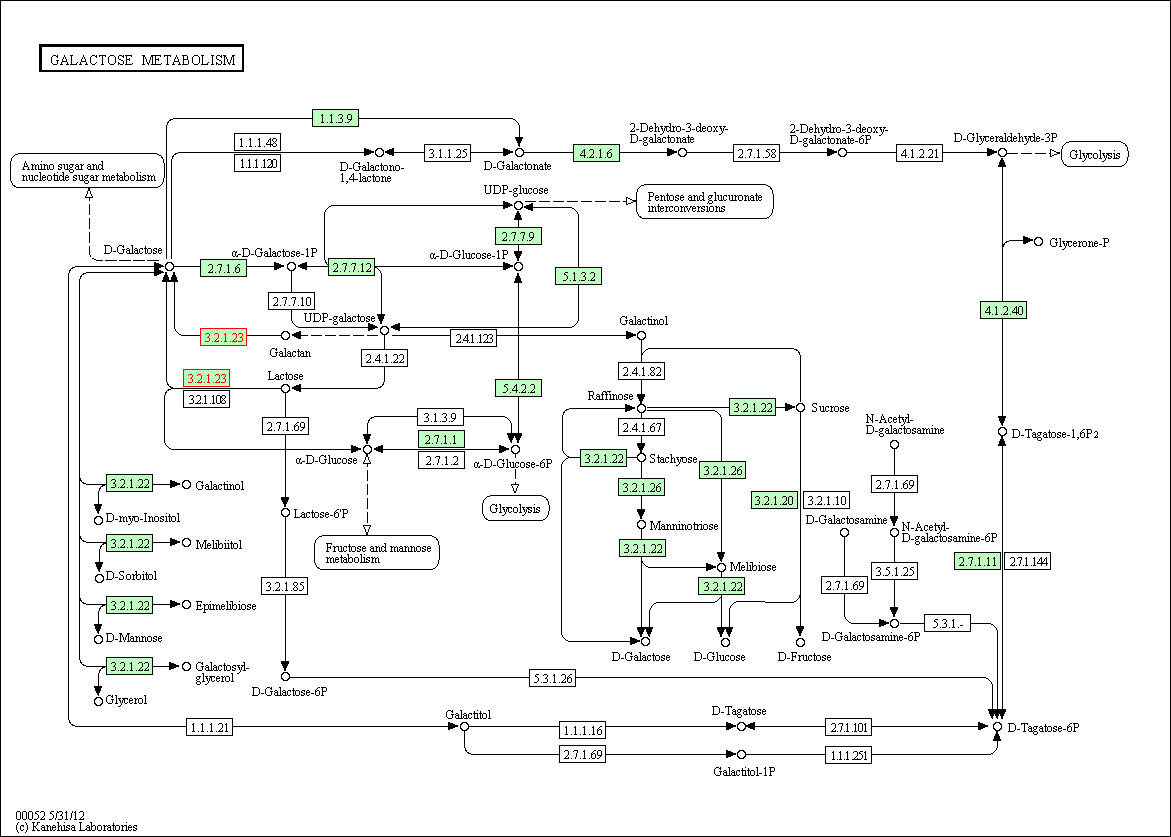
Beta-galaktozydaza uczestniczy w metabolizmie galaktozy. W bazie KEGG znalazłam schemat tego szlaku metabolicznego. Do tego celu użyłam Bazy KEGG organisms. Wśród organizmów znalazłam Penicilium chrysogenum, a następnie wpisałam w okno wyszukiwania ‚beta galactosidase'. Na schemacie enzym ten oznaczony jest numerem dostępowym EC 3.2.1.23 (żółta ramka). Bierze udział w dwóch przemianach tj. przemianie galaktanu do D-galaktozy oraz przemianie laktozy do D-galaktozy.
U bakterii Escherichia coli K12 MG1655 również występuje szlak metaboliczny glukozy (metoda poszukiwania jak wyżej). Drożdże Saccharomyces cerevisiae nie uczestniczą jednak w tym szlaku. Nie mogą wykorzystywać laktozy jako źródło węgla ponieważ nie wytwarzają beta-galaktozydazy (laktazy), która rozkłada laktozę do galaktozy i glukozy.
Ćwiczenie 2
Po wprowadzeniu hasła „beta galactosidase” do zakładki szukaj znajduję w bazie danych CATH informacje przedstawione w tabeli.
W bazie SCOP po wprowadzeniu hasła ”beta- galactosidase” znalazłam dla Penicillium sp. informacje przedstawione w tabeli (dla domeny 2). Wybrałam domenę 2, aby porównać ją z domeną 2 w bazie CATH.
Program |
Klasyfikacja strukturalna |
Budowa |
Funkcje biologiczne |
CATH |
--> 2v4vA00[Author:K] głównie struktura beta, Rodzaj fałdowania: kanapka |
Składa się z dwóch domen
Główną domeną reprezentatywną jest: domena 2-1tg7A02, zawiera głównie strukturę beta, 3-warstwowa kanapka.
|
Baza CATH łączy się z bazą PDB, a w PDB znalazłam następujące funkcje: katabolizm węglowodanów i polisacharydów, procesy metaboliczne |
SCOP |
Beta-galactosidase LacA, domena 2 -głównie struktura beta Domena 2: Rodzaj fałdowania: Pofałdowana kartka
|
(oprócz domeny 2 enzym ma również domenę 3, 4 i 5)
|
metabolizm węglowodanów, degradacja polisacharydów (link Q700S9 przekierował do bazy UniProtKB) |
Baza danych SCOP zawiera wyniki klasyfikacji białek przeprowadzonej na podstawie badania zależności ewolucyjnych i strukturalnych, natomiast CATH dobiera wyniki tylko na podstawie struktury drugorzędowej. Zarówno baza SCOP jak i CATH, przy klasyfikacji wykorzystują strukturę drugorzędową. Według obu baz, w badanym enzymie występuje głównie struktura beta. Różnią się one jednak rodzajem fałdowania domeny 2. Według SCOP jest to pofałdowana kartka, a według CATH jest to 3-warstwowa kanapka. CATH i SCOP są bazami hierarchicznymi. Nie pokazują więc bezpośrednio funkcji biologicznych. Przekierowują natomiast do bazy PDB i UniProtKB, a te bazy pokazują takie same funkcje --> biologiczne[Author:K] .
Trochę krótko…
pI
co to znaczy?
Brakuje mi opisu wyników (lub ich wyszczególnienia) na każdym z etapów klasyfikacji z obydwu bazach.
Wyszukiwarka
Podobne podstrony:
Orzechowski Michał lab 2, MGR, sem I, Bioinformatyka zaawansowana, L4 Inne bazy danych białkowych sz
RAPORT - LAB 4 - Agata Jackiewicz - sprawdzony, MGR, sem I, Bioinformatyka zaawansowana
adresy Pozyskiwanie funduszy unijnych 15.03.2011 Sz.D, Studia Meil Energetyka, MGR, SEM 3, INTERGRAC
ZESTAWIENIE STALI 11 01 15, Polibuda mgr, SEM III, konst. metalowe, Konstrukcje metalowe, stale proj
Organizacja Laboratorium Usługowego1B, studia-biologia, Studia magisterskie, Mgr sem III, Diagnostyk
Zarządzanie wiedzą, studia-biologia, Studia magisterskie, Mgr sem III, Zarządzanie wiedzą
CWICZENIE PROJEKTOWE 11 01 15, Polibuda mgr, SEM III, konst. metalowe, Konstrukcje metalowe, stale p
pytania od doswiadczonej koleżanki -), Polibuda mgr, SEM III, konst. metalowe, Konstrukcje metalowe,
spis tresci, Polibuda mgr, SEM III, konst. metalowe, Konstrukcje metalowe, stale projekt 11 01 15
mgr Piotr Greniewski - zadania sem.4(1), ewsie, Bazy Danych
Wyk+Ă©ady[1], Biotechnologia 3 rok 1 i 2 sem, Bioinformatyka
Fitoplankton, studia-biologia, Studia magisterskie, Mgr sem II, Hydrobiologia, na koło do goni raryt
Organizacja Laboratorium Usługowego1A, studia-biologia, Studia magisterskie, Mgr sem III, Diagnostyk
Met Spek Biotech 2012, MGR, sem I, Metody spektroskopowe
Ściąga na pierwsze koło, MGR, sem I, Innowacje w Biotechnologii
STAL SEM III, Polibuda mgr, SEM III, konst. metalowe, Konstrukcje metalowe, stale egzamin
więcej podobnych podstron