Sprawozdanie z zajęć ćwiczeniowych z agrobiotechnologii
Temat ćwiczeń:
Analiza restrykcyjna bakteriofaga λ
Cel ćwiczeń:
Strawienie DNA bakteriofaga λ oraz analiza wyników w oparciu o mapy restrykcyjne badanych enzymów.
Zasada metody:
Pierwszym krokiem w analizie sekwencji sklonowanego odcinka DNA jest ustalenie, jak uszeregowane są na nim miejsca cięcia enzymami restrykcyjnymi. Enzymy restrykcyjne przecinają duże cząsteczki DNA, dając powtarzalne zbiory stosunkowo niewielkich fragmentów DNA. Fragmenty te można rozdzielić ze względu na ich wielkość dzięki elektroforezie w półpłynnym żelu. Ogólnie rzecz biorąc, im krótszy jest taki dwuniciowy odcinek, tym szybciej porusza się on w polu elektrycznym. Stosując różne rodzaje elektroforezy, można rozdzielać fragmenty liczące od dwudziestu do milionów par zasad. Używając wzorców, czyli odcinków o znanej wielkości, można łatwo określić wielkość badanego fragmentu. Do przeprowadzenia analizy wystarcza zwykle mniej niż mikrogram (milionowa część grama) DNA, ponieważ odcinki DNA można łatwo wykryć po wybarwieniu ich odpowiednim barwnikiem. Zestaw fragmentów powstających w wyniku działania określonej restryktazy stanowi swoisty „odcisk palca” wyjściowego DNA. Analizując fragmenty powstałe w wyniku działania różnych enzymów restrykcyjnych oraz ich rozmaitych kombinacji, można często ustalić kolejność występowania badanych fragmentów w wyjściowej cząsteczce DNA. W rezultacie otrzymujemy fizyczną mapę DNA, która, jeśli jest wystarczająco szczegółowa, jednoznacznie opisuje oryginalną cząsteczkę DNA. Zrekombinowane wektory są zwykle niewielkie, skonstruowanie ich mapy fizycznej nie przedstawia więc dużych trudności. Jednak trawienie enzymami restrykcyjnymi dużych cząsteczek DNA daje złożone zestawy fragmentów.
Enzymy restrykcyjne - enzymy z grupy endonukleaz przecinające nić DNA w miejscu wyznaczanym przez specyficzną sekwencję DNA. Rozpoznawana sekwencja z reguły ma charakter symetryczny o długości od 4 do 8 par zasad (pz), choć zdarzają się częste wyjątki. Restryktazy wraz z metylazami DNA stanowią system restrykcji i modyfikacji DNA, który w organizmach prokariotycznych stanowi mechanizm odpornościowy zapobiegający infekcjom przez bakteriofagi.
Podział enzymów restrykcyjnych:
Typy enzymów:
Typ I - wielopodjednostkowe kompleksy enzymatyczne zawierające aktywności metylazy i restryktazy. Przecinają DNA z dala od rozpoznawanej sekwencji, w bliżej nieokreślonym miejscu. Z tego powodu nie mają większego zastosowania praktycznego.
Typ II - przecinają DNA w zdefiniowanym miejscu, w obszarze rozpoznawanej sekwencji lub w jej pobliżu. Składają się z pojedynczych polipeptydów. Rozpoznają sekwencje symetryczne.
Typ IIs - zbliżone do typu II, przecinają z jednej strony rozpoznawanej sekwencji, która jest asymetryczna.
Typ III - duże kompleksy, które wymagają dwóch sekwencji rozpoznawanych w pobliżu siebie. Bez znaczenia praktycznego.
Typ IV - zbliżone do typu II. Zawierają aktywność metylazy i restryktazy w tym samym polipeptydzie. Przecinają DNA w zdefiniowanym obszarze poza sekwencją rozpoznawania. Aktywności metylazy i restryktazy nie mogą działać równocześnie. Enzym "przełącza" się w zależności od substratu.
Podział według rodzaju wytwarzanych końców:
tępe końce - nici rozcięte naprzeciwko siebie - wszystkie nukleotydy są sparowane z komplementarnymi nukleotydami na przeciwnym łańcuchu
lepkie końce - 3` lub 5` ssDNA (jednoniciowe) ogony na obu końcach utworzone przez niesymetryczne cięcie , komplementarne do podobnych tworzonych w innych cząsteczkach DNA przez te same enzymy restrykcyjne niezależnie od źródła DNA , co pozwala na ligowanie DNA nawet bardzo różniących się gatunków czyli formowanie chimerycznych molekuł.
Podział według sekwencji rozpoznawanej:
czwórkowe - rozpoznają sekwencję DNA złożoną z czterech nukleotydów. Statystycznie w dowolnym DNA takich miejsc jest dużo - co 256 bp. Restryktazy takie mogą strawić DNA na bardzo małe kawałki.
szóstkowe - rozpoznają sekwencję DNA złożoną z sześciu nukleotydów. Dowolne miejsce restrykcyjne złożone z sześciu nukleotydów występuje statystycznie co około 4096 bp w DNA , w którym ilości poszczególnych nukleotydów są równe. Proporcje te zmieniają się w zależności od organizmu , dlatego dobór enzymu szóstkowego i warunki należy ustalić eksperymentalnie.
ósemkowe - stosowane niezbyt często. Tną DNA bardzo rzadko.
Nomenklatury enzymów użytych podczas ćwiczeń:
BamH I - pierwszy enzym wyizolowany z Bacillus amyloliquefaciens szczepu H
EcoRI - pierwszy enzym wyizolowany z Escherichia coli szczepu R
Hind III - trzeci enzym wyizolowany z Haemophilus influenzae szczepu d
Sal I - pierwszy enzym wyizolowany z Streptomyces albus
Bgl II - drugi enzym wyizolowany z Bacillus globigii
Enzym |
Rozpoznawana sekwencja |
Cięcie |
EcoRI |
5'GAATTC |
5'---G AATTC---3' |
|
3'CTTAAG |
3'---CTTAA G---5' |
|
|
|
BamH I |
5'GGATCC |
5'---G GATCC---3' |
|
3'CCTAGG |
3'---CCTAG G---5' |
|
|
|
Hind III |
5'AAGCTT |
5'---A AGCTT---3' |
|
3'TTCGAA |
3'---TTCGA A---5' |
|
|
|
Sal I |
5'GTCGAC |
5'---G TCGAC---3' |
|
3'CAGCTG |
3'---CAGCT G---5' |
|
|
|
Bgl II |
5'AGATCT |
5'---A GATCT---3' |
|
3'TCTAGA |
3'---TCTAG A---5' |
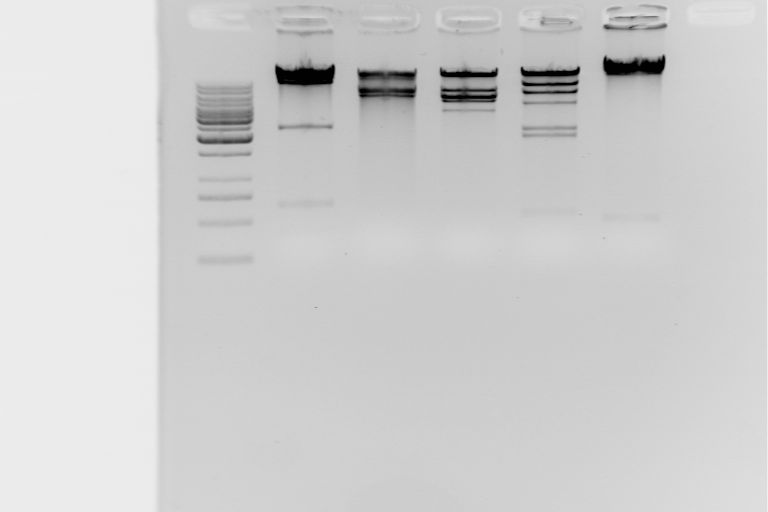
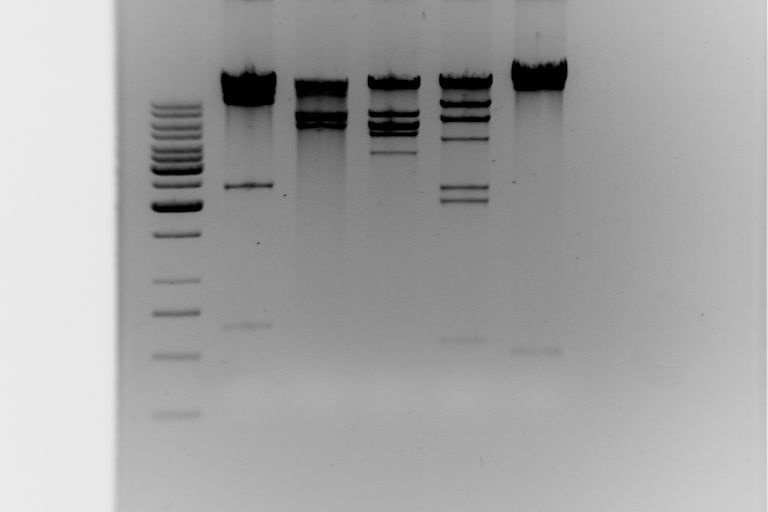
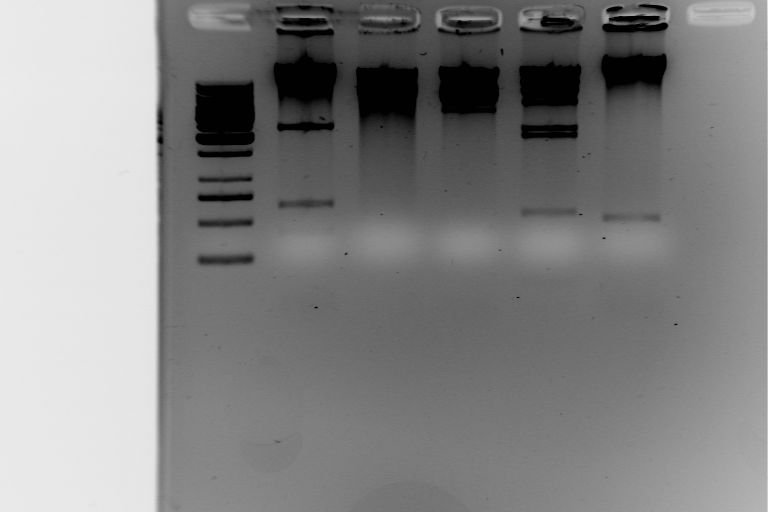
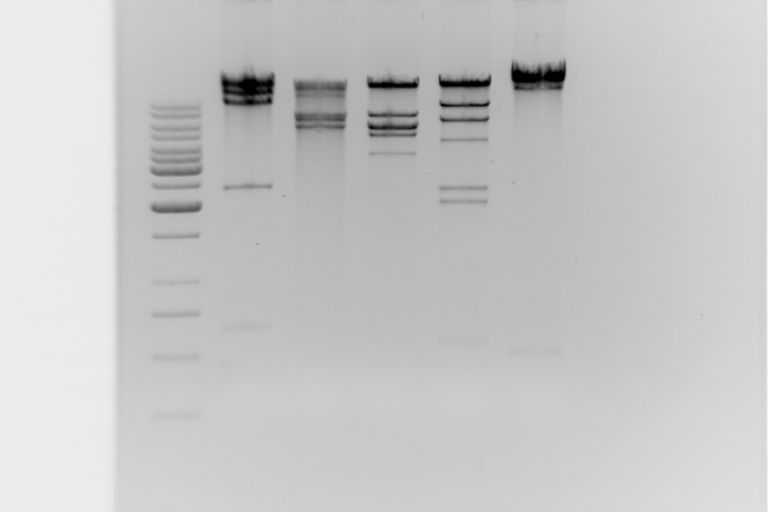
Wyniki:
W 1 studzience zastosowaliśmy marker 1kb DNA. Jest jednym z najpopularniejszych markerów, pozwalających określić wielkość dwuniciowego DNA o masie od 250 do 10000 bp. Fragmenty wielkości 1000 i 3000 bp na żelu agarozowym barwionym bromkiem etydyny są widoczne z relatywnie większą intensywnością w porównaniu do innych prążków i służą jako punkt odniesienia.

Wnioski:
Analiza długości fragmentów restrykcyjnych jest metodą inżynierii genetycznej wykorzystującą polimorfizm długości fragmentów restrykcyjnych (RFLP). W metodzie tej wykorzystuje się trawienie DNA za pomocą enzymów restrykcyjnych i elektroforezę. Już zmiana pojedynczego nukleotydu w sekwencji rozpoznawanej przez enzym spowoduje, że nie dokona on rozcięcia DNA, co powoduje, że np. w dwóch różnych próbkach powstanie różna ilość fragmentów DNA, będących produktami rozkładu pierwotnej cząsteczki. Fragmenty te będą się także różnić wielkością. Elektroforeza umożliwi rozróżnienie wielkości tych fragmentów, co będzie widoczne jako różnica w ilości i położeniu poszczególnych prążków na elektroforogramie.
W naszym doświadczeniu można odczytać następujące długości prążków na 5 zdjęciach powstałe w wyniku cięcia enzymami restrykcyjnymi:
Enzym BglII - 700
2600
8000
12000
BamH I -7000
9000
12000
EcoR I - 4500
5500
6500
9000
13000
Hind III - 2100
2400
7000
8000
11000
13000
Sal I - 2000
11000
Uzyskane wyniki można porównać z wirtualnym strawieniem DNA bakteriofaga lambda:
Bgl II
# |
Ends |
Coordinates |
Length (bp) |
1 |
BglII-BglII |
416-22425 |
22010 |
2 |
BglII-BglII |
22426-35711 |
13286 |
3 |
BglII-(RightEnd) |
38815-48502 |
9688 |
4 |
BglII-BglII |
35712-38103 |
2392 |
5 |
BglII-BglII |
38104-38754 |
651 |
6 |
(LeftEnd)-BglII |
1-415 |
415 |
7 |
BglII-BglII |
38755-38814 |
60 |
|
|
|
|
||
|
||
BamHI
# |
Ends |
Coordinates |
Length (bp) |
1 |
BamHI-BamHI |
5506-22346 |
16841 |
2 |
BamHI-BamHI |
34500-41732 |
7233 |
3 |
BamHI-(RightEnd) |
41733-48502 |
6770 |
4 |
BamHI-BamHI |
27973-34499 |
6527 |
5 |
BamHI-BamHI |
22347-27972 |
5626 |
6 |
(LeftEnd)-BamHI |
1-5505 |
5505 |
EcoRI
|
|
# |
Ends |
Coordinates |
Length (bp) |
1 |
(LeftEnd)-EcoRI |
1-21226 |
21226 |
2 |
EcoRI-EcoRI |
31748-39168 |
7421 |
3 |
EcoRI-EcoRI |
39169-44972 |
5804 |
4 |
EcoRI-EcoRI |
26105-31747 |
5643 |
5 |
EcoRI-EcoRI |
21227-26104 |
4878 |
6 |
EcoRI-(RightEnd) |
44973-48502 |
3530 |
HindIII
# |
Ends |
Coordinates |
Length (bp) |
1 |
(LeftEnd)-HindIII |
1-23130 |
23130 |
2 |
HindIII-HindIII |
27480-36895 |
9416 |
3 |
HindIII-HindIII |
37460-44141 |
6682 |
4 |
HindIII-(RightEnd) |
44142-48502 |
4361 |
5 |
HindIII-HindIII |
25158-27479 |
2322 |
6 |
HindIII-HindIII |
23131-25157 |
2027 |
7 |
HindIII-HindIII |
36896-37459 |
5 |
SalI
# |
Ends |
Coordinates |
Length (bp) |
1 |
(LeftEnd)-SalI |
1-32745 |
32745 |
2 |
SalI-(RightEnd) |
33245-48502 |
15258 |
3 |
SalI-SalI |
32746-33244 |
49 |
Sal I
EcoR I
Hind III
10000
8000
6000
5000
4000
3500
3000
2500
2000
1500
250
500
750
1000
BamH I
Marker
Bgl II
Wyszukiwarka
Podobne podstrony:
enzymy restrykcyjne
ENZYMY RESTRYKCYJNe
1001562-enzymy restrykcyjne, semestr IV, genetyka, Genetyka
Inżynieria genetyczna enzymy restrykcyjne
enzymy restrykcyjne-stud, Studia, Inżynieria genetyczna
enzymy restrykcyjne-stud, Biologia molekularna
Enzymy restrykcyjne
Enzymy restrykcyjne
1 Trawienie wektora enzymy restrykcyjne
enzymy restrykcyjne
Enzymy restrykcyjne i reakcja łańcuchowa polimerazy(PCR) zastosowanie (2)
enzymy restrykcyjne
Wykład 1 Enzymy restrykcyjne
enzymy
Referat Inżynieria Produkcji Rolniczej
pros 4 Enzymy 1
więcej podobnych podstron