
64
www.postepybiochemii.pl
Sylwester Głowacki
Janusz Błasiak
Katedra Genetyki Molekularnej, Wydział
Biologii i Ochrony Środowiska, Uniwersytet
Łódzki, Łódź
Katedra Genetyki Molekularnej, Wydział
Biologii i Ochrony Środowiska, Uniwersytet
Łódzki, ul. Pomorska 141/143, 90-236 Łódź; e-
-mail sglowa@biol.uni.lodz.pl
Artykuł otrzymano 16 listopada 2012 r.
Artykuł zaakceptowano 14 lutego 2013 r.
Słowa kluczowe: metylacja DNA, epigenety-
ka, 5-hydroksymetylocytozyna, regulacja eks-
presji genów
Wykaz skrótów: 5-caC — 5-karboksycytozyna;
5-fC — 5-formylcytozyna; 5-hmC — 5-hydro-
ksymetylocytozyna; 5-mC — 5-metylocytozy-
na
Rola 5-hydroksymetylocytozyny i białek Tet
w epigenetycznej regulacji ekspresji genów
STRESZCZENIE
M
etylacja DNA pełni ważną rolę w epigenetycznej regulacji ekspresji genów człowieka.
Przez długi czas było kwestią dyskusyjną, czy w komórkach ssaków istnieją mecha-
nizmy aktywnej demetylacji DNA. Niedawne odkrycie białek rodziny Tet, zdolnych prze-
kształcać 5-metylocytozynę w 5-hydroksymetylocytozynę, pozwoliło opisać szlak, w którym
dochodzi do aktywnej demetylacji DNA. Wyniki dalszych badań sugerują, że 5-hydroksy-
metylocytozyna jest nie tylko produktem przejściowym w procesie demetylacji DNA, ale
może także regulować profil epigenetyczny genomu człowieka.
WPROWADZENIE
Metylacja DNA jest jedną z najważniejszych modyfikacji epigenetycznych ge-
nomu człowieka. Nie zmienia ona sekwencji DNA, jest jednak zachowywana
w trakcie podziałów komórkowych. Substratem dla metylacji DNA są cytozy-
ny wchodzące w skład dinukleotydów CpG, których większość zgromadzona
jest w obrębie wysp CpG, charakterystycznych dla obszarów regulatorowych
genomów ssaków, a produktem 5-metylocytozyna (5-mC). Metylacja DNA w
promotorach genów może prowadzić do wyciszenia ich ekspresji [1]. Z kolei
utrata metylacji może prowadzić do hamowania wzrostu i apoptozy, zarówno w
komórkach prawidłowych jak i nowotworowych [2,3]. Metylacja DNA odgrywa
także ważną rolę w rozwoju embrionalnym, co potwierdzają wyniki badań
przeprowadzonych na myszach [4]. Metylacja DNA może być przeprowadzana
de novo przez metylotransferazy DNA DNMT3a i DNMT3b lub odtwarzać wzo-
rzec metylacji po replikacji przez metylazę DNMT1. Przez długi czas jedynym
powszechnie akceptowanym mechanizmem demetylacji DNA w komórkach
ssaków była pasywna demetylacja będąca skutkiem podziałów komórkowych,
prowadzących do rozrzedzania wzoru metylacji. Jednakże do demetylacji DNA
dochodzi także w komórkach nieprzechodzących cytokinez, w których nie za-
chodzi replikacja DNA [5]. Choć u roślin obserwuje się proces demetylacji po-
wodowany działaniem glikozylaz DNA, enzymy te wykazują znacznie słabszą
aktywność w stosunku do 5-mC ssaków [6].
5-hydroksymetylocytozyna (5-hmC), stosunkowo niedawno odkryta mody-
fikacja cytozyny, powstaje w reakcji enzymatycznej oksydacji 5-mC przepro-
wadzanej przez enzym Tet1 (ang. ten-eleven translocation 1) (Tab. 1) [7]. Dopro-
wadziło to do wysunięcia hipotezy, zgodnie z którą 5-hmC jest produktem po-
średnim reakcji prowadzących do usuwania metylocytozyny z DNA [8]. 5-mC
charakteryzuje się wysoką stabilnością chemiczną. Utlenienie tego związku
do 5-hmC usuwa grupę metylową, ale pozostawia egzocykliczny podstawnik
Tabela 1. Historia badań nad 5-hmC i białkami Tet.
Data
Wydarzenie
Piśmiennictwo
1952
identyfikacja 5-hmC w DNA bakteriofagów
[11]
1972
pierwsza praca opisująca wysoki poziom 5-hmC w
komórkach ssaków; brak niezależnego potwierdzenia
tych wyników doprowadził do zarzucenia
badań nad 5-hmC na wiele kolejnych lat
[12]
2003
wskazanie, że Tet1 jest składnikiem fuzyjnej
onkoproteiny w ostrej białaczce szpikowej
[13]
2009
wykrycie 5-hmC w komórkach Purkiniego i mózgu, oraz
wykazanie, że Tet1 dokonuje oksydacji 5-mC do 5-hmC
[7,14]
2010
zwrócono uwagę, że wyniki dotyczące bisulfidowania
powinny być zweryfikowane, gdyż 5-hmC zachowuje
się przy zastosowaniu tej techniki tak samo jak 5-mC
[14]

Postępy Biochemii 59 (1) 2013
65
w pozycji 5 [9]. Ponieważ jednak 5-hmC powstała z 5-mC,
blokuje ona wiązanie się z DNA enzymu DNTM1 i odtwa-
rzanie wzoru metylacji po replikacji DNA, przekształcenie
5-mC do 5-hmC może być traktowane jako zniesienie znacz-
nika epigenetycznego [9,10]. Dalsze badania z wykorzysta-
niem profilowania genomowego wykazały jednak, że roz-
kład 5-hmC odbiega od rozkładu 5-mC i 5-hmC znajduje się
w promotorach wielu genów, co sugeruje, że związek ten
funkcjonować może także jako samodzielny epigenetyczny
regulator ekspresji genów o funkcji odmiennej od 5-mC [8].
BIOGENEZA 5-hmC I ROLA BIAŁEK Tet
Białka z rodziny Tet zawierającej trzy paralogi to zależ-
ne od oksoglutaranów i żelaza dioksygenazy (Tab. 2) [16].
U myszy produkty wszystkich trzech genów mogą prze-
kształcać 5-mC w 5-hmC. Poziom metylacji DNA wzrasta w
wyniku wyciszenia aktywności genów Tet [17]. Co więcej,
enzymy te katalizować mogą także inne przemiany 5-mC w
5-formylcytozynę (5-fC) i 5-karboksycytozynę (5-caC), zaś
obie te pochodne wykryte zostały w DNA embrionalnych
komórek macierzystych (ESC) oraz w pełni zróżnicowa-
nych komórek myszy [18].
Białka Tet zawierają wśród swoich domen bogate w resz-
ty cysteiny rejony odpowiedzialne za wiązanie DNA. Tet1 i
Tet3 zawierają także dodatkowe domeny zawierające palce
cynkowe CXXC, podobne do domen wiążących DNA, wy-
stępujących w DNMT1. Z drugiej strony analiza genomu
wskazuje, że domena ta w przypadku genu Tet2 zachowa-
ła się jako osobne białko, którego gen został oddzielony od
oryginalnego genu Tet2 w wyniku inwersji chromosomowej
[16]. Nie wiadomo jeszcze, czy cechy strukturalne tej dome-
ny w Tet1 umożliwiają, czy wręcz przeciwnie, uniemożli-
wiają wiązanie się tego białka z DNA [19,20]. Wyniki badań
sugerują, że domena ta nie bierze udziału w wiązaniu DNA
i jest zbędna dla zachowania katalitycznej aktywności Tet1
in vivo [19]. Zasugerowano, że aktywne wiązanie się z DNA
poprzez domeny zawierające palce cynkowe może bloko-
wać dostęp metylotransferaz DNA i w ten sposób dodat-
kowo negatywnie modulować poziom metylacji DNA [20].
W Tet1 zidentyfikowano też unikalne, bogate w reszty cy-
steiny struktury podobne do występujących w białku AlkB,
gdzie funkcjonują jako domeny wiążące DNA [16]. Ponadto
w białkach Tet zidentyfikowano domeny mogące ulegać su-
moilacji, co wskazuje, że modyfikacja ta może być jednym
ze sposobów na regulację aktywności tych białek [16].
ROLA 5-hmC W METABOLIZMIE KOMóRKOWYM
5-hmC może funkcjonować jako produkt pośredni w
szlakach demetylacji DNA. W tym celu musi zostać pod-
dana dalszym przemianom, które doprowadzą do odtwo-
rzenia niezmetylowanej cytozyny w miejscu pierwotnie
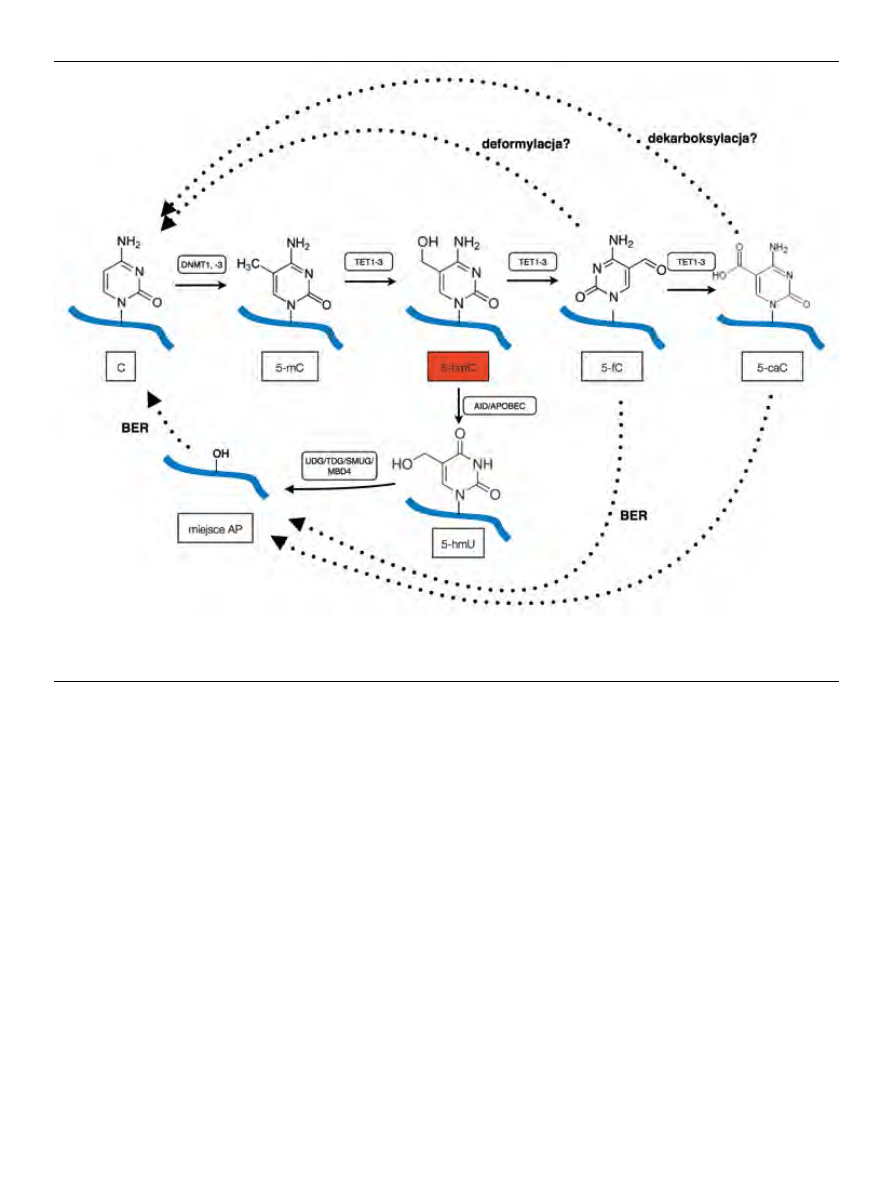
występującej 5-mC (Ryc. 1). Jednym z możliwych szlaków
jest szlak deaminacji z wykorzystaniem aktywności deami-
nazy AID/APOBEC, na co wskazywałaby korelacja między
nadprodukcją AID/APOBEC a spadkiem stężenia 5-hmC
[5]. Jednoczesna nadprodukcja Tet1 i AID/APOBEC pro-
wadzi z kolei do akumulacji 5-hydroksymetylouracylu,
deaminowanej pochodnej 5-hmC, która usuwana jest przez
glikozylazę DNA uracylu 1 [5,21]. W szlaku tym mogą tak-
że brać udział białko MBD4, które wykazuje zdolność usu-
wania 5-hydroksyuracylu; oraz glikozylaza tyminy w DNA
(TDG), usuwająca tyminę z błędnego sparowania G:T [22].
TDG może też usuwać oksydacyjne pochodne 5-hmC takie
jak 5-fC i 5-caC [22]. Pochodne te mogą być też usuwane
w szlaku naprawy DNA przez wycinanie zasad azotowych
(BER), choć sugeruje się także, że ich przemiana zachodzić
może również na drodze deformylacji i dekarboksylacji
[8,23].
Dotychczas biologiczna rola 5-hmC była najdokładniej
badana w kontekście rozwoju osobniczego, w regulacji pro-
cesów krwiotwórczych, roli w funkcjonowaniu komórek
nerwowych oraz w nowotworach [8]. W warunkach in vi-
tro w kulturach komórek z nadprodukcją Tet1 stwierdzono
obniżoną ilość 5-mC i nieznacznie podwyższoną ilość nie-
zmetylowanej cytozyny [7]. W dojrzałych tkankach poziom
5-hmC jest zróżnicowany i zwiera się w przedziale 0,03-
0,69%, osiągając największe wartości w neuronach, w któ-
rych stanowi do około 0,7% wszystkich cytozyn. Poziom ten
jest niższy w rejonach hipokampa bogatych w komórki ma-
cierzyste [24]. Z drugiej strony, wyniki badań nad komór-
kami macierzystymi myszy sugerują, że wyciszenie genów
Tet1 i Tet2 wywołuje spadek aktywności genów związanych
z pluripotencją i wzrost poziomu metylacji ich promotorów.
Sugerowałoby to, że poziom hydroksymetylacji DNA spa-
da wraz z różnicowaniem się komórek [25]. Badano także
wpływ procesów metylacji i demetylacji DNA na liczbę ko-
pii mtDNA w komórkach różnych tkanek. Zaobserwowano
na przykład, że 5-hmC jest elementem systemów regulują-
cych syntezę kodowanej jądrowo polimerazy DNA γ, której
aktywność istotnie wpływa na liczbę kopii mtDNA [26].
Tabela 2. Charakterystyka ludzkich enzymów Tet. Dane pochodzą z bazy uniprot.org.
Cecha
Tet1
Tet2
Tet3
Długość (aminokwasy)
2136
2002
1660
Masa (Da)
235 309
223 811
179 350
Katalizowana reakcja
DNA 5mC + 2-oksoglutaran + O
2
→ DNA 5-hmC + bursztynian + CO
2
Kofaktor
1Fe
2+
na podjednostkę
Specyficzność tkankowa
syntezowany w sercach, płucach i
mózgu płodów, ale nie wykryty w
tych samych narządach u dorosłych;
wykryty w mięśniach szkieletowych,
grasicy i jajnikach dorosłych
syntezowany w wielu tkankach, szczególnie
mocna w komórkach hematopoetycznych, przy
czym najwyższa ekspresja obserwowana jest
w granulocytach; ekspresja w granulocytach
krwi obwodowej zredukowana u pacjentów
cierpiących na zespoły mielodysplastyczne
synteza w jelicie,
mięśniach i
limfocytach krwi
obwodowej
66
www.postepybiochemii.pl
Należy także zaznaczyć, że 5-hmC może działać jak in-
hibitor metylacji zachowawczej, hamując wiązanie się z
hemimetylowanymi sekwencjami enzymu DNMT1. 5-hmC
powstająca jako produkt oksydacji 5-mC przez reaktywne
formy tlenu, pośredniczy w ten sposób w akumulacji zabu-
rzeń profilu metylacji DNA indukowanych stresem oksyda-
cyjnym [27]. Oddziaływanie między IDH (dehydrogenaza
izocytrynianu) a Tet sugeruje istnienie relacji pomiędzy
stresem środowiskowym a hydroksymetylacją DNA. Za-
proponowano mechanizm, w którym stres oksydacyjny in-
dukuje aktywację SIRT3, które z kolei aktywuje IDH2. Wy-
tworzony przez IDH2 α-ketoglutaran aktywuje Tet, które
przetwarzają 5-mC do 5-hmC [28].
ROLA 5-hmC I BIAŁEK Tet W REGULOWANIU ROZWOJU
Aktywność Tet ma fundamentalne znaczenie dla pra-
widłowego rozwoju zarodkowego. Poszczególne enzymy
z rodziny Tet są aktywne na różnych etapach rozwoju i/
lub w różnych tkankach. Tet1 jest głównym enzymem ak-
tywnym w ESC, zaś wyciszenie jego aktywności na drodze
interferencji RNA jest pozytywnie skorelowane ze spad-
kiem zawartości 5-hmC w genomie tych komórek [7]. Tet3
osiąga najwyższą aktywność w oocytach i zygotach, gdzie
najprawdopodobniej uczestniczy w reprogramowaniu ge-
nomu ojcowskiego po zapłodnieniu. Proces ten powiązany
jest ze znacznym wzrostem poziomu 5-hmC i ustaje po osią-
gnięciu stadium dwukomórkowego [29]. Jak dotąd jednak
nie wykazano bezpośrednio enzymatycznej aktywności
Tet3 w katalizowaniu przemiany 5-mC w 5-hmC [8]. Nie-
mniej proces reprogramowania epigenetycznego w klono-
waniu reprodukcyjnym zwierząt wymaga aktywności Tet3
i jest analogiczny do reprogramowania ojcowskich projąder
po zapłodnieniu [30].
Drugim obok reprogramowania epigenetycznego proce-
sem związanym z rozwojem jest piętnowanie genetyczne,
czyli zjawisko specyficznego zachowania rodzicielskiego
wzoru metylacji w wybranych genach. Geny poddane pięt-
nowaniu muszą być zabezpieczone przed aktywną demety-
lacją przeprowadzaną przez białka Tet. Jeden z wykrytych
mechanizmów działa w oparciu o aktywność białka STEL-
LA. Mutanty z nieaktywną formą tego białka nie rozróż-
niają w trakcie pierwszych etapów rozwoju zarodkowego
genomu matczynego i ojcowskiego i dokonują aktywnej
demetylacji obu, w tym elementów retrotranspozonowych
zawartych w genomie matczynym. Prowadzi to do zaha-
mowania rozwoju zarodkowego na wczesnym etapie. Wy-
kazano, że specyficzna rekrutacja STELLA do sekwencji
Rycina 1. Rola 5-hydroksymetylocytozyny (5-hmC) w szlakach metylacji i demetylacji DNA. Metylacja DNA przeprowadzana jest przez metylotransferazy DNA(DNMT).
Powstała 5-metylocytozyna (5-mC) jest modyfikowana przez enzymy z rodziny TET do 5-hmC i jej dalszych pochodnych. 5-hmC może być deaminowana do 5-hydrok-
symetylouracylu (5-hmU), usuwanego następnie w szlaku naprawy DNA przez wycinanie zasad azotowych. C — cytozyna; 5-fC — 5-formylocytozyna; 5-caC — 5-kar-
boksycytozyna. Na podstawie [9], zmienione.

Postępy Biochemii 59 (1) 2013
67
poddanych piętnowaniu hamuje wiązanie białek Tet i w ten
sposób chroni je przed demetylacją [31].
Wyniki badań z zastosowaniem immunofluorescencji
sugerują, że 5-hmC znajduje się zazwyczaj w obszarach eu-
chromatyny, w tym z rejonami oddziałującymi z sekwen-
cjami wzmacniającymi (ang. enhancers) i wykazującymi
metylację H3K4me1 i H3K27ac [32]. Stwierdzono także, że
poziom 5-hmC w mysich embrionalnych komórkach macie-
rzystych jest podwyższony w regionach wiążących czynni-
ki transkrypcyjne związane z pluripotencjalnością, zaś wy-
ciszenie Tet1 i Tet2 skutkuje ich metylacją i hamowaniem
ekspresji [25,33]. Choć nie obniża to żywotności, myszy z
delecją Tet2 wykazują zmiany rozwojowe i tendencję do no-
wotworów krwi lub szpiku [34]. Jednym z mechanizmów
takiego działania może być osłabianie przez 5-hmC oddzia-
ływania z czynnikami wiążącymi metylowane DNA [35].
Jednakże obecność mutacji Tet1 w embrionalnych komór-
kach macierzystych myszy ma mniejszy wpływ na rozwój
zarodkowy [17,36]. Choć wyciszenie genów Tet wywołuje
hamowanie ekspresji szeregu genów, powoduje ono także
uaktywnienie innych, w tym będących obiektem działania
kompleksu represorowego Polycomb 2 [37]. Sugeruje to po-
dwójną rolę jaką może pełnić 5-hmC, zarówno aktywatora
i represora ekspresji genów. Wśród indukowanych genów
znajduje się szereg markerów różnicowania, w tym Cdx2,
Eomes, Gata4 i Gata6 [38].
ROLA 5-hmC I BIAŁEK Tet W HEMATOPOEZIE
I NOWOTWORZENIU
Mutacje Tet2 wpływające na jego aktywność katalitycz-
ną są częste u chorych na białaczki różnego typu, zaś ich
obecność jest skorelowana z obniżonym poziomem 5-hmC
[39]. Zauważono, że w komórkach niektórych białaczek do-
chodzi do obniżenia aktywności białek Tet i jednoczesnego
spadku poziomu metylacji DNA. Zaproponowano, iż może
to wynikać z zaburzenia mechanizmów regulujących mety-
lację DNA [39]. Sugeruje się, że mutacje Tet2 mogą odgrywać
rolę w regulacji aktywności komórek macierzystych krwi i
procesach ich różnicowania. Myszy z mutacją Tet2 zmienia-
jącą aktywność domeny katalitycznej charakteryzowały się
podwyższonym poziomem hematopoetycznych komórek
macierzystych krwi (HSC, ang. hematopoietic stem cells). W
testach transplantacyjnych, HSC z mutacją w Tet2 wykazały
selekcyjną przewagę nad prawidłowymi HSC [40]. Myszy z
obniżoną aktywnością Tet2 rozwijają się początkowo pra-
widłowo, lecz z czasem zaczynają zapadać na różne choro-
by układu krwiotwórczego, w tym obejmujące zaburzenia
różnicowania się komórek [37,41]. Myszy z ekspresją Tet2
zredukowaną o 80% umierają już po trzech dniach od naro-
dzin, zaś w ich wątrobach obserwuje się wysokie poziomu
komórek progenitorowych krwi [42]. Jedną z konsekwencji
utraty aktywności Tet2 jest wzrost potencjału komórek ma-
cierzystych do odnawiania się. Co więcej, właściwości takie
obserwuje się nawet w komórkach z wyłączoną tylko jedną
kopią Tet2 (Tet2
+/-
) [41]. Stwierdzono także oddziaływanie
między konstytutywnie aktywną onkogenną kinazą tyro-
zynową BCR/ABL a białkami Tet. Oddziaływanie to pro-
wadzi do umiejscowienia Tet w cytozolu i spadku poziomu
5-hmC w genomie. Oddziaływanie inhibitora BCR/ABL
imatynibu, z kinazą BCR/ABL reaktywuje Tet i przywraca
fizjologiczny poziom 5-hmC w DNA [43]. Na rolę białek Tet
w hematopoezie wskazują także oddziaływania z innymi
białkami. Mutacje w genach białek IDH1 i IDH2 występują
często u pacjentów cierpiących na ostrą białaczkę szpikową.
Produktem aktywności IDH jest α-ketoglutaran. Stwier-
dzono, że zmutowane warianty tych białek upośledzają
aktywność Tet2 i hamują różnicowanie się komórek w trak-
cie hematopoezy [44]. Zwrócono także uwagę na potencjał
diagnostyczny i prognostyczny utraty hydroksymetylacji w
rozwoju czerniaka. Proces ten związany jest z utratą aktyw-
ności dehydrogenazy 2 izocytrynianu oraz enzymów Tet. W
badaniach na zwierzętach stwierdzono, że wprowadzenie
aktywnych form Tet osłabia wzrost nowotworu i zwiększa
przeżywalność zwierząt doświadczalnych [45].
ROLA 5-hmC I BIAŁEK Tet W TKANKACH NERWOWYCH
Tkanki nerwowe były obiektem badań, które pozwoliły
zrekonstruować niektóre z hipotetycznych szlaków deme-
tylacji opisanych wcześniej. Jako komórki niedzielące się,
stały się kandydatem do poszukiwania szlaków aktywnej
demetylacji, ponieważ w zróżnicowanych neuronach nie
zachodzi pasywna demetylacja na drodze podziału ko-
mórkowego [5]. W przypadku badań nad tkanką nerwową
myszy stwierdzono, że poziom 5-hmC może rosnąć wraz
z rozwojem i różnicowaniem komórek macierzystych ukła-
du nerwowego u dorosłych myszy [46]. Z wynikami tymi
jest spójna korelacja stwierdzona między stopniem rozwo-
ju móżdżku u człowieka a poziomem akumulacji 5-hmC w
jego tkankach [47]. Jest to także zgodne ze stwierdzonymi
obniżonymi poziomami 5-hmC pozytywnie skorelowany-
mi z anaplazjami w guzach mózgu [48]. Pod tym względem
funkcja 5-hmC wydaje się być w komórkach mózgu inna
niż w komórkach macierzystych, gdzie zwykle różnicowa-
niu towarzyszy spadek ilości 5-hmC. Różnice dotyczą także
rozkładu 5-hmC, w komórkach nerwowych jest ona raczej
nieobecna w regionach promotorowych, występuje zaś czę-
ściej w regionach kodujących genów [49]. Zmiany w hy-
droksymetylacji mogą też odgrywać rolę w rozwoju chorób
neurologicznych. Stwierdzono zwiększoną częstość zmian
w hydroksymetylacji genów powiązanych z rozwojem au-
tyzmu [47]. Wykazano, że pewne formy autyzmu związane
są z mutacjami w genie białka MeCP2, które w warunkach
in vitro blokuje aktywnością Tet1 [9].
Jak się wydaje 5-hmC i enzymy z rodziny Tet uczestniczą
także w epigenetycznej regulacji ekspresji genów mitochon-
drialnych w tkankach mózgu [50]. Stwierdzono obecność
5-hmC w DNA mitochondrialnym, a także lokalizację bia-
łek Tet w mitochondriach. Zauważono także, że wpływ na
aktywność Tet i poziom 5-hmC może mieć proces starze-
nia [50]. W móżdżku myszy w szeregu genów dochodzi do
akumulacji 5-hmC w trakcie starzenia, przy czym geny te
są związane z takimi procesami jak podatność na hipoksję,
angiogeneza czy związane z wiekiem procesy neurodege-
neracyjne [9].
PODSUMOWANIE
Odkrycie aktywności TET pogłębiło naszą wiedzę na
temat demetylacji DNA, która jest jednym z istotnych me-
chanizmów kontroli aktywności genów na poziomie epi-

68
www.postepybiochemii.pl
genetycznym. W ostatnich kilku latach uzyskano szereg
wyników potwierdzających znaczenie hydroksymetylacji
w regulacji takich procesów jak rozwój i różnicowanie ko-
mórek macierzystych, procesy transformacji nowotworowej
czy procesy starzenia. Jednakże pełna rola 5-hmC pozostaje
do wyjaśnienia. Dalszych badań wymaga określenie różnic
między rolą 5-hmC w regulacji fizjologii komórek macierzy-
stych, a rolą w komórkach zróżnicowanych. Także szlaki
przekształceń 5-hmC prowadzące do odtworzenia niezme-
tylowanej cytozyny wymagają dalszych badań. Wstępne
wyniki wskazują także, że badania nad 5-hmC mogą do-
starczyć nowych informacji o genezie i rozwoju niektórych
chorób.
PIŚMIENNICTWO
1. Prokhortchouk E, Defossez P-A (2008) The cell biology of DNA meth-
ylation in mammals. Biochim Biophys Acta 1783: 2167-2173
2. Jackson-Grusby L, Beard C, Possemato R, Tudor M, Fambrough D,
Csankovszki G, Dausman J, Lee P, Wilson C, Lander E, Jaenisch R
(2001) Loss of genomic methylation causes p53-dependent apoptosis
and epigenetic deregulation. Nat Genet 27: 31-39
3. Chen T, Hevi S, Gay F, Tsujimoto N, He T, Zhang B, Ueda Y, Li E
(2007) Complete inactivation of DNMT1 leads to mitotic catastrophe
in human cancer cells. Nat Genet 39: 391-396
4. Takebayashi S-I, Tamura T, Matsuoka C, Okano M (2007) Major and
essential role for the DNA methylation mark in mouse embryogenesis
and stable association of DNMT1 with newly replicated regions. Mol
Cell Biol 27: 8243-8258
5. Guo JU, Su Y, Zhong C, Ming G-L, Song H (2011) Hydroxylation of
5-methylcytosine by TET1 promotes active DNA demethylation in the
adult brain. Cell 145: 423-434
6. Zhu J-K (2009) Active DNA demethylation mediated by DNA glyco-
sylases. Annu Rev Genet 43: 143-166
7. Tahiliani M, Koh KP, Shen Y, Pastor WA, Bandukwala H, Brudno Y,
Agarwal S, Iyer LM, Liu DR, Aravind L, Rao A (2009) Conversion of
5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by
MLL partner TET1. Science 324: 930-935
8. Branco MR, Ficz G, Reik W (2012) Uncovering the role of 5-hydroxy-
methylcytosine in the epigenome. Nat Rev Genet 13: 7-13
9. Kriukienė E, Liutkevičiūtė Z, Klimašauskas S (2012) 5-Hydroxymeth-
ylcytosine — the elusive epigenetic mark in mammalian DNA. Chem
Soc Rev 41: 6916-6930
10. Inoue A, Zhang Y (2011) Replication-dependent loss of 5-hydroxy-
methylcytosine in mouse preimplantation embryos. Science 334: 194
11. Wyatt GR, Cohen SS (1952) A New Pyrimidine Base from Bacterio-
phage Nucleic Acids. Nature 170: 1072-1073
12. Penn NW, Suwalski R, O’Riley C, Bojanowski K, Yura R (1972) The
presence of 5-hydroxymethylcytosine in animal deoxyribonucleic
acid. Biochem J 126: 781-790
13. Lorsbach RB, Moore J, Mathew S, Raimondi SC, Mukatira ST, Down-
ing JR (2003) TET1, a member of a novel protein family, is fused to
MLL in acute myeloid leukemia containing the t(10;11)(q22;q23). Leu-
kemia 17: 637-641
14. Kriaucionis S, Heintz N (2009) The nuclear DNA base 5-hydroxymeth-
ylcytosine is present in Purkinje neurons and the brain. Science 324:
929-930
15. Jin S-G, Kadam S, Pfeifer GP (2010) Examination of the specificity of
DNA methylation profiling techniques towards 5-methylcytosine and
5-hydroxymethylcytosine. Nucleic Acids Res 38: e125
16. Iyer LM, Tahiliani M, Rao A, Aravind L (2009) Prediction of novel
families of enzymes involved in oxidative and other complex modifi-
cations of bases in nucleic acids. Cell Cycle 8: 1698-1710
17. Ito S, D’Alessio AC, Taranova OV, Hong K, Sowers LC, Zhang Y (2010)
Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal
and inner cell mass specification. Nature 466: 1129-1133
18. Ito S, Shen L, Dai Q, Wu SC, Collins LB, Swenberg JA, He C, Zhang Y
(2011) Tet proteins can convert 5-methylcytosine to 5-formylcytosine
and 5-carboxylcytosine. Science 333: 1300-1303
19. Frauer C, Rottach A, Meilinger D, Bultmann S, Fellinger K, Hasenöder
S, Wang M, Qin W, Söding J, Spada F, Leonhardt H (2011) Differ-
ent binding properties and function of CXXC zinc finger domains in
Dnmt1 and Tet1. PLoS ONE 6: e16627
20. Xu Y, Wu F, Tan L, Kong L, Xiong L, Deng J, Barbera AJ, Zheng L,
Zhang H, Huang S, Min J, Nicholson T, Chen T, Xu G, Shi Y, Zhang
K, Shi YG (2011) Genome-wide regulation of 5hmC, 5mC, and gene
expression by Tet1 hydroxylase in mouse embryonic stem cells. Mol
Cell 42: 451-464
21. Cortellino S, Xu J, Sannai M, Moore R, Caretti E, Cigliano A, Le Coz M,
Devarajan K, Wessels A, Soprano D, Abramowitz LK, Bartolomei MS,
Rambow F, Bassi MR, Bruno T, Fanciulli M, Renner C, Klein-Szanto
AJ, Matsumoto Y, Kobi D, Davidson I, Alberti C, Larue L, Bellacosa A
(2011) Thymine DNA glycosylase is essential for active DNA demeth-
ylation by linked deamination-base excision repair. Cell 146: 67-79
22. Sjolund AB, Senejani AG, Sweasy JB (2012) MBD4 and TDG: Multifac-
eted DNA glycosylases with ever expanding biological roles. Mutat
Res. doi: 10.1016/j.mrfmmm.2012.11.001
23. Maiti A, Drohat AC (2011) Thymine DNA glycosylase can rapidly ex-
cise 5-formylcytosine and 5-carboxylcytosine: potential implications
for active demethylation of CpG sites. J Biol Chem 286: 35334-35338
24. Globisch D, Münzel M, Müller M, Michalakis S, Wagner M, Koch S,
Brückl T, Biel M, Carell T (2010) Tissue distribution of 5-hydroxymeth-
ylcytosine and search for active demethylation intermediates. PLoS
ONE 5: e15367
25. Ficz G, Branco MR, Seisenberger S, Santos F, Krueger F, Hore TA,
Marques CJ, Andrews S, Reik W (2011) Dynamic regulation of 5-hy-
droxymethylcytosine in mouse ES cells and during differentiation.
Nature 473: 398-402
26. Kelly RDW, Mahmud A, McKenzie M, Trounce IA, St John JC (2012)
Mitochondrial DNA copy number is regulated in a tissue specific man-
ner by DNA methylation of the nuclear-encoded DNA polymerase
gamma A. Nucleic Acids Res 40: 10124-10138
27. Valinluck V, Sowers LC (2007) Endogenous cytosine damage products
alter the site selectivity of human DNA maintenance methyltransfer-
ase DNMT1. Cancer Res 67: 946-950
28. Chia N, Wang L, Lu X, Senut M-C, Brenner C, Ruden DM (2011) Hy-
pothesis: environmental regulation of 5-hydroxymethylcytosine by
oxidative stress. Epigenetics 6: 853-856
29. Iqbal K, Jin S-G, Pfeifer GP, Szabó PE (2011) Reprogramming of the
paternal genome upon fertilization involves genome-wide oxidation
of 5-methylcytosine. Proc Natl Acad Sci USA 108: 3642-3647
30. Gu T-P, Guo F, Yang H, Wu H-P, Xu G-F, Liu W, Xie Z-G, Shi L, He X,
Jin S, Iqbal K, Shi YG, Deng Z, Szabó PE, Pfeifer GP, Li J, Xu G-L (2011)
The role of Tet3 DNA dioxygenase in epigenetic reprogramming by
oocytes. Nature 477: 606-610
31. Messerschmidt DM (2012) Should I stay or should I go: protection and
maintenance of DNA methylation at imprinted genes. Epigenetics 7:
969-975
32. Szulwach KE, Li X, Li Y, Chun-Xiao Song C-X, Han JW, Kim SS, Nam-
buri S, Hermetz K, Kim JJ, Rudd MK, Yoon Y-S, Ren B, He C, Jin P
(2011) Integrating 5-hydroxymethylcytosine into the epigenomic land-
scape of human embryonic stem cells. PLoS Genet 7: e1002154
33. Wu H, D’Alessio AC, Ito S, Wang Z, Cui K, Zhao K, Sun YE, Zhang
Y (2011) Genome-wide analysis of 5-hydroxymethylcytosine distribu-
tion reveals its dual function in transcriptional regulation in mouse
embryonic stem cells. Genes Dev 25: 679-684
34. Li Z, Cai X, Cai C-L, Wang J, Zhang W, Petersen BE, Yang F-C, Xu
M (2011) Deletion of Tet2 in mice leads to dysregulated hematopoi-
etic stem cells and subsequent development of myeloid malignancies.
Blood 118: 4509-4518
35. Valinluck V, Tsai H-H, Rogstad DK, Burdzy A, Bird A, Sowers LC
(2004) Oxidative damage to methyl-CpG sequences inhibits the bind-
ing of the methyl-CpG binding domain (MBD) of methyl-CpG binding
protein 2 (MeCP2). Nucleic Acids Res 32: 4100-4108
Postępy Biochemii 59 (1) 2013
69
36. Dawlaty MM, Ganz K, Powell BE, Hu Y-C, Markoulaki S, Cheng AW,
Gao Q, Kim J, Choi S-W, Page DC, Jaenisch R (2011) Tet1 is dispens-
able for maintaining pluripotency and its loss is compatible with em-
bryonic and postnatal development. Cell Stem Cell 9: 166-175
37. Quivoron C, Couronné L, Valle Della V, Lopez CK, Plo I, Wagner-Bal-
lon O, Cruzeiro MD, Delhommeau F, Arnulf B, Stern M-H, Godley L,
Opolon P, Tilly H, Solary E, Duffourd Y, Philippe Dessen, Merle-Beral
H, Nguyen-Khac F, Fontenay M, Vainchenker W, Bastard C, Mercher
T, Bernard OA (2011) TET2 inactivation results in pleiotropic hemato-
poietic abnormalities in mouse and is a recurrent event during human
lymphomagenesis. Cancer Cell 20: 25-38
38. Wu H, D’Alessio AC, Ito S, Xia K, Wang Z, Cui K, Zhao K, Sun YE,
Zhang Y (2011) Dual functions of Tet1 in transcriptional regulation in
mouse embryonic stem cells. Nature 473: 389-393
39. Ko M, Huang Y, Jankowska AM, Pape UJ, Tahiliani M, Bandukwa-
la HS, An J, Lamperti ED, Koh KP, Ganetzky R, Liu XS, Aravind L,
Agarwal S, Maciejewski JP, Rao A (2010) Impaired hydroxylation of
5-methylcytosine in myeloid cancers with mutant TET2. Nature 468:
839-843
40. Ko M, Bandukwala HS, An J, Lamperti ED, Thompson EC, Hastie R,
Tsangaratou A, Rajewsky K, Koralov SB, Rao A (2011) Ten-Eleven-
Translocation 2 (TET2) negatively regulates homeostasis and differen-
tiation of hematopoietic stem cells in mice. Proc Natl Acad Sci USA
108: 14566-14571
41. Moran-Crusio K, Reavie L, Shih A, Abdel-Wahab O, Ndiaye-Lobry D,
Lobry C, Figueroa ME, Vasanthakumar A, Patel J, Zhao X, Perna F,
Pandey S, Madzo J, Song C, Dai Q, He C, Ibrahim S, Beran M, Zavadil
J, D Nimer SD, Melnick A, Godley LA, Aifantis I, Levine RL (2011)
Tet2 loss leads to increased hematopoietic stem cell self-renewal and
myeloid transformation. Cancer Cell 20: 11-24
42. Shide K, Kameda T, Shimoda H, Yamaji T, Abe H, Kamiunten A,
Sekine M, Hidaka T, Katayose K, Kubuki Y, Yamamoto S, Miike T,
Iwakiri H, Hasuike S, Nagata K, Marutsuka K, Iwama A, Matsuda T,
Kitanaka A, Shimoda K (2012) TET2 is essential for survival and hema-
topoietic stem cell homeostasis. Leukemia 26: 2216-2223
43. Mancini M, Veljkovic N, Leo E, Aluigi M, Borsi E, Galloni C, Iacobucci
I, Barbieri E, Santucci MA (2012) Cytoplasmatic compartmentalization
by Bcr-Abl promotes TET2 loss-of-function in chronic myeloid leuke-
mia. J Cell Biochem 113: 2765-2774
44. Figueroa ME, Abdel-Wahab O, Lu C, Ward PS, Patel J, Shih A, Li Y,
Bhagwat N, Vasanthakumar A, Fernandez HF, Tallman MS, Sun Z,
Wolniak K, Peeters JK, Liu W, Choe SE, Fantin VR, Paietta E, Löwen-
berg B, Licht JD, Godley LA, Delwel R, Valk PJM, Thompson CB,
Levine RL, Melnick A (2010) Leukemic IDH1 and IDH2 mutations
result in a hypermethylation phenotype, disrupt TET2 function, and
impair hematopoietic differentiation. Cancer Cell 18: 553-567
45. Lian CG, Xu Y, Ceol C, Wu F, Larson A, Dresser K, Xu W, Tan L, Hu
Y, Zhan Q, Lee C-W, Hu D, Lian BQ, Kleffel S, Yang Y, Neiswender J,
Khorasani AJ, Fang R, Lezcano C, Duncan LM, Scolyer RA, Thompson
JF, Kakavand H, Houvras Y, Zon LI, Mihm MC, Kaiser UB, Schatton
T, Woda BA, Murphy GF, Shi YG (2012) Loss of 5-hydroxymethylcyto-
sine is an epigenetic hallmark of melanoma. Cell 150: 1135-1146
46. Song C-X, Szulwach KE, Fu Y, Dai Q, Yi C, Li X, Li Y, Chen C-H, Zhang
W, Jian X, Wang J, Zhang L, Looney TJ, Zhang B, Godley LA, Hicks
LM, Lahn BT, Jin P, He C (2011) Selective chemical labeling reveals
the genome-wide distribution of 5-hydroxymethylcytosine. Nat Bio-
technol 29: 68-72
47. Wang T, Pan Q, Lin L, Szulwach KE, Song C-X, He C, Wu H, Warren
ST, Jin P, Duan R, Li X (2012) Genome-wide DNA hydroxymethyl-
ation changes are associated with neurodevelopmental genes in the
developing human cerebellum. Hum Mol Genet. doi: 10.1093/hmg/
dds394
48. Kraus TFJ, Globisch D, Wagner M, Eigenbrod S, Widmann D, Münzel
M, Müller M, Pfaffeneder T, Hackner B, Feiden W, Ulrich Schüller U,
Carell T, Kretzschmar HA (2012) Low values of 5-hydroxymethylcyto-
sine (5hmC), the “sixth base”, are associated with anaplasia in human
brain tumors. Int J Cancer 131: 1577-1590
49. Szulwach KE, Li X, Li Y, Song C-X, Wu H, Dai Q, Irier H, Upadhyay
AK, Gearing M, Levey AI, Vasanthakumar A, Godley LA, Chang
Q, Cheng X, He C, Jin P (2011) 5-hmC-mediated epigenetic dynam-
ics during postnatal neurodevelopment and aging. Nat Neurosci 14:
1607-1616
50. Dzitoyeva S, Chen H, Manev H (2012) Effect of aging on 5-hydroxy-
methylcytosine in brain mitochondria. Neurobiol Aging 33: 2881-2891
Role of 5-hydroxymethylcytosine and Tet proteins in
epigenetic regulation of gene expression
Sylwester Glowacki
, Janusz Blasiak
Department of Molecular Genetics, University of Lodz, 141/143 Pomorska St., 90-236 Lodz, Poland
e-mail sglowa@biol.uni.lodz.pl
Key words: DNA methylation, 5-hydroksymethylcytosine, epigenetics, gene expression regulation
ABSTRACT
DNA methylation plays an important role in epigenetic regulation of human gene expression. Mechanism of active demethylation of the
human genome have been a matter of discussion for many years. Recently, a novel group of TET protein family enzymatically converting
5-methylcytosine into 5-hydroxymethylcytosine was discovered, playing a role in active DNA demethylation pathway. Results obtained in
subsequent studies pointed that 5-hydroxymethylcytosine was not only an intermediate in that pathway, but might also modify epigenetic
profile of the human genome.
Wyszukiwarka
Podobne podstrony:
05 med dosw 4 2013id 5960 Nieznany (2)
05 PO Filtracja 2013id 5967 Nieznany (2)
05 med dosw 4 2013id 5960 Nieznany (2)
05 Majid 5744 Nieznany (2)
cw PAiTS 05 id 122324 Nieznany
2015 04 09 08 25 05 01id 28644 Nieznany (2)
NAI2006 05 id 313056 Nieznany
05 Spoinyid 5835 Nieznany
matma dyskretna 05 id 287941 Nieznany
cwiczenie 05 id 125057 Nieznany
05 Wykonywanie, odczytywanie i Nieznany
05 Occhid 5768 Nieznany (2)
714[01] Z1 05 Malowanie farba e Nieznany
05 TSid 5852 Nieznany (2)
05 Wodaid 5865 Nieznany (2)
pp test odp zima 05 air boratyn Nieznany
lab pwsp 05 id 258618 Nieznany
1 rok mgr22,02,2013id 9707 Nieznany
03 PO Geometria 2013id 4609 Nieznany (2)
więcej podobnych podstron